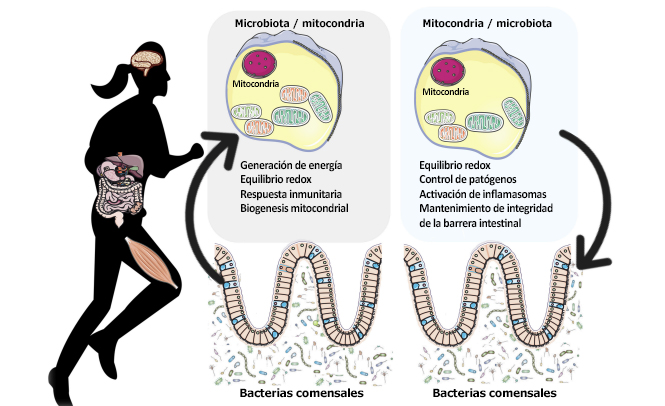
05 Ago Comunicación Bidireccional entre la Microbiota Intestinal y las Mitocondrias durante el Ejercicio
Allison Clark1 y Núria Mach 1, 2
1 Departamento de Ciencias de la Salud, Universidad Abierta de Cataluña, Barcelona, España.
2 UMR 1313, INRA, AgroParisTech, Université Paris-Saclay, Jouy-en-Josas, Francia.
En respuesta al ejercicio de resistencia se producen numerosos cambios fisiológicos que permiten la adaptación a las crecientes necesidades de energía; biogénesis de mitocondrias, incremento en la producción de especies reactivas de oxígeno (ROS) y respuestas inflamatorias agudas. Las mitocondrias son organelas intracelulares que son fundamentales para la producción de ATP y que también son importantes productoras de ROS y especies reactivas de nitrógeno durante el ejercicio de alta intensidad. Evidencia reciente demuestra que existe una interacción bidireccional entre las mitocondrias y la microbiota. Se ha demostrado que la microbiota intestinal regula coactivadores transcripcionales claves, factores de transcripción y enzimas que intervienen en la biogénesis mitocondrial tales como PGC-1α, SIRT1 y los genes de la enzima AMPK. Además, la microbiota intestinal y sus metabolitos, como los ácidos grasos de cadena corta y los ácidos biliares secundarios, también participan en la producción de energía del huésped, en la modulación de las ROS y en la inflamación en el intestino al atenuar la respuesta inmune mediada por TNFα y los inflamasomas como NLRP3. Por otro lado, las mitocondrias, y particularmente la producción mitocondrial de ROS, tienen un papel crucial en la regulación de la microbiota intestinal a través de la modulación del funcionamiento de la barrera intestinal y de la respuesta inmune de la mucosa. Recientemente, también se ha demostrado que las variantes genéticas que existen dentro del genoma mitocondrial, podrían afectar la función mitocondrial y, por lo tanto, la composición y la actividad de la microbiota intestinal. También se sabe que la dieta modula dramáticamente la composición de la microbiota intestinal. Por lo tanto, los estudios que se realicen sobre la microbiota intestinal pueden ser útiles para controlar la producción de ROS asociada a mitocondrias, las señales proinflamatorias y los límites metabólicos en atletas de resistencia.
Palabras clave: microbiota intestinal, energía, resistencia, inflamación, mitocondrias, estrés oxidativo.
Abreviaturas: 8-OhdG=8-hidroxi-2-desoxiguanosina; ACC= Acetil CoA carboxilasa; AP-1= proteína activadora 1; AMP= Adenosin monofosfato; ATP= Adenosin trifosfato; AMPK= proteína quinasa activada por AMP; ANGTPL4= similar a la angiopoyetina 4; atp1=ATP sintasa 1; atp3=ATP sintasa 3; ChREBP, proteína de unión al elemento sensible a carbohidratos; AMPc=adenosin monofosfato cíclico; CREB=proteína de unión al elemento sensible a AMP cíclico; COX= citocromo c oxidasa; FXR= receptor farnesoide X; FIAF= factor adiposo inducido por el ayuno; FAO= β-oxidación de ácidos grasos; FFAR2= receptor de ácidos grasos libres 2; FFAR3=receptor de ácidos grasos libres 3; GPR= receptor de proteína acoplada a G; TGR5= proteína de membrana 5 acoplada a G; GF= libre de gérmenes; GPx=glutatión peroxidasa; GSH-Px= glutatión peroxidas; H2S=sulfuro de hidrógeno; HPA=hipotálamo-hipófisis suprarrenal; IL-6=interleuquina 6; LPS=lipopolisacárido; Mn-SOD=manganeso superóxido dismutasa; NAD=dinucleótido de nicotinamida y adenina; NADP= NAD fosfato; NO=óxido nítrico; NLRP3= familia de receptores similares a NOD que contiene un dominio pirina 3; NF-κB=factor nuclear kappa B; NRF1=factor respiratorio nuclear 1; NRF2=factor respiratorio nuclear 2; OXPHOS, fosforilación oxidativa; GSSG=glutatión oxidado; PGC-1α=proteína 1α coactivadora del receptor activado por el proliferador de peroxisomas; PPARγ= receptor gama activado por proliferador de peroxisoma; ROS=especies reactivas de oxígeno; RONS=especies reactivas de nitrógeno y oxígeno; GSH=glutatión reducido; SCFA= ácido graso de cadena corta; SIRT1= regulador silencioso 1; SPF=específico libre de patógenos; STAT3= transductor de señal y activador de transcripción 3; SREBP-1c, proteína de unión al elemento regulador del esterol 1c; SOD=superóxido dismutasa; TLR4=receptor tipo toll 4; TCA=ácido tricarboxílicos; T3=triyodotironina (receptor 43); TNFα=factor de necrosis tumoral alfa; UCP2=proteína de desacoplamiento 2.
INTRODUCCIÓN
El ejercicio de resistencia puede ser definido como aquel ejercicio cardiovascular (correr, practicar esquí campo traviesa, ciclismo, ejercicio aeróbico o natación), que se realiza durante un período prolongado (Joyner y Coyle, 2008; Mach y Fuster-Botella, en prensa) El ejercicio de resistencia requiere una gran cantidad de adaptaciones fisiológicas para cumplir con las demandas de energía y para mantener la homeostasis corporal (Mach y Fuster-Botella, en prensa). Los principales cambios fisiológicos que se producen durante el ejercicio de alta intensidad incluyen: (i) contracciones musculares coordinadas (Spriet y Watt, 2003), (ii) oxidación de glucosa y ácidos grasos (Spriet y Watt , 2003), (iii) incremento en el consumo de las reservas de glucagón (Spriet y Watt , 2003), (iv) fosforilación oxidativa (Befroy et al, 2008), (v) biogénesis mitocondrial en diferentes tejidos, incluidos los músculos (Radak et al. , 2008), (vi) balance de electrolitos y de la temperatura, (vii) aumento de la producción de especies reactivas de oxígeno (ROS) y de especies reactivas de oxígeno y nitrógeno (RONS), vii) activación de los ejes simpato-adreno- medular e hipotálamo-hipófisis-adrenal (HPA), lo que produce la liberación de hormonas del estrés al sistema circulatorio (revisado por Clark y Mach, 2016) así como también inflamación sistémica y respuestas inmunitarias (Mach y Fuster-Botella, en prensa). En algunos casos, la hipoxia y la hipoperfusión gastrointestinal aumentan la permeabilidad intestinal y el estrés oxidativo en el tracto gastrointestinal (Magalhães y col. 2013; Clark y Mach, 2016).
El rol que desempeñan las mitocondrias durante el ejercicio de resistencia va mucho más allá de su capacidad de producción de energía. Las células de los mamíferos contienen cientos a miles de mitocondrias y el tamaño, forma y cantidad de las mismas dependerá de las diferentes condiciones y de los estímulos fisiológicos tales como el ejercicio de resistencia, las altas temperaturas, la dieta y las hormonas (Bartlett y Eaton , 2004; Knuiman et al., 2015; Busquets-Cortés et al., 2016). Las mitocondrias son organelas que son los centros de energía primaria, y en ellas se oxidan las grasas y los azúcares para generar adenosin trifosfato (ATP). La fosforilación oxidativa mitocondrial (OXPHOS), que combina el transporte de electrones con la respiración celular y la síntesis de ATP (Lee y Wei, 2005; Cheng y Ristow, 2013) y la β oxidación de ácidos grasos (FAO) son las dos vías metabólicas que son centrales para estos procesos. Las mitocondrias también pueden usar vías enzimáticas del ciclo de los ácidos tricarboxílicos (TCA) para generar ATP (aproximadamente 20% de ATP; Papa y col. 2012). Estas organelas también están involucradas en otros procesos metabólicos y celulares esenciales, entre los que se incluyen la homeostasis de calcio, la señalización intracelular, la biosíntesis del grupo hemo, y la muerte celular aguda (Wai y Langer, 2016). Como producto secundario de la respiración normal, las mitocondrias producen ROS y RONS reactivas (Green y col. 2011), que tienen roles importantes en las vías de señalización y homeostasis celular, pero una cantidad excesiva de ROS puede además provocar daños significativos en las estructuras celulares y la inducción de citoquinas o muerte celular por apoptosis (Green y col. 2011).
Además, a medida que se replican, sus genomas acumulan mutaciones que eventualmente comprometen la eficiencia de la OXPHOS (Green et al. 2011). Las mitocondrias también juegan un papel central en la iniciación de la inflamación a través de inflamasomas, un conjunto molecular de funciones que activan la caspasa-1, lo que facilita la secreción de las citoquinas inflamatorias IL-1, IL-18 y de otros mediadores de la inflamación (Green y col. 2011).
Está claro que las funciones mitocondriales son importantes durante las actividades metabólicas demandantes tales como el ejercicio de resistencia. En comparación con otros atletas, los atletas de resistencia tienen una mayor cantidad y volumen de mitocondrias en el músculo esquelético para cubrir las necesidades energéticas (Befroy y col. 2008; Hood et al., 2011; Busquets- Cortés et al., 2016). Se ha observado un aumento en la biogénesis para mejorar el rendimiento de resistencia muscular gracias a su mayor capacidad para realizar OXPHOS y β-oxidación de ácidos grasos o de cuerpos cetónicos y, por lo tanto, de producción de energía (Hood y col. 2011).
Se ha demostrado que en atletas de resistencia, la producción moderada de ROS y RONS estimula la biogénesis mitocondrial y la FAO (Wai y Langer, 2016). Sin embargo, los desequilibrios redox durante períodos prolongados de tiempo se han asociado con un inicio rápido de fatiga, con incapacidad de mantener la velocidad y la intensidad de rendimiento (Rapoport, 2010). Además, el ejercicio extenuante provoca un aumento en la cantidad de citoquinas proinflamatorias, tales como TNFα, IL-1, IL-6, moduladores antiinflamatorios y proteína 1 inflamatoria de macrófagos, lo que indica un efecto de dosis-respuesta entre las respuestas biológicas al ejercicio y la inmunidad del huésped (revisado por Mach y Fuster-Botella, en prensa). Debido al rol fundamental de las mitocondrias en la activación de los inflamasomas y en otras respuestas inflamatorias, se ha puesto especial atención en las mitocondrias durante el ejercicio de resistencia.
Una nueva investigación demuestra que existe una comunicación bidireccional entre la microbiota intestinal y las mitocondrias (Ma J. y col. 2014; Mottawea et al., 2016; Saint-Georges-Chaumet y Edeas, 2016). La microbiota intestinal contiene más de 100 billones de microorganismos (Rajilic-Stojanovic y de Vos , 2014), que pertenecen aproximadamente a 160 especies y poseen aprox. 9 millones de genes (Li y col. 2014). La microbiota intestinal es clave para el metabolismo del huésped ya que participa en la digestión y absorción de alimentos (Neis y col. 2015), neutraliza medicamentos y carcinógenos, sintetiza colina ( Nicholson et al., 2012), ácidos biliares secundarios ( Hylemon et al., 2009; Sagar et al. 2015 ; Joyce y Gahn, 2016), folato ( Sugahara et al., 2015), vitamina K2 ( Marley et al. , 1986) y ácidos grasos de cadena corta (SCFA). Además, la microbiota intestinal protege al huésped contra infecciones de patógenos (Lozupone y col. 2012), estimula y madura el sistema inmunitario (Vighi y col. 2008) y las células epiteliales (Hooper y Gordon, 2001) y regula el estrés oxidativo (Xu y col. 2014).
La interacción entre microbiota y mitocondrias se produciría principalmente a través de la señalización desde la microbiota intestinal hacia las mitocondrias y desde las mitocondrias hacia la microbiota intestinal a través de vínculos endocrinos, inmunitarios y humorales (Mottawea y col. 2016). La evidencia más directa de las interacciones mitocondria-microbiota provienen de estudios sobre funciones mitocondriales que se ven afectadas durante infecciones bacterianas, y sobre las diferentes estrategias desarrolladas por patógenos bacterianos para alterar funciones relacionadas con la homeostasis del calcio, el mantenimiento del estado redox y la morfología mitocondrial (revisado por Lobet et al. , 2015). Los patobiontes (i.e, Fusobacterium, Veillonella y Atopobium parvulum) tienden a controlar la actividad mitocondrial a favor de la infección y de la inflamación a través de la producción de sulfuro de hidrógeno (H2S) y óxido nítrico (NO) (Mottawea y col. 2016). Otros estudios recientes demostraron que los metabolitos producidos por la microbiota intestinal comensal, entre los que se incluyen los beneficiosos SCFA y ácidos biliares secundarios, pueden influir en las funciones mitocondriales relacionadas con la producción de energía, la biogénesis mitocondrial, el equilibrio redox y las cascadas inflamatorias, lo que la convierte en un potencial objetivo terapéutico para la resistencia (Circu y Aw, 2012; Bär y cols. 2013; den Besten et al., 2013; Mottawea et al., 2016). Por ejemplo, la microbiota intestinal comensal reduce la producción de ROS a través de los SCFA como el N-butirato (Mottawea y col. 2016).
Por otro lado, las funciones mitocondriales pueden modificar la composición y actividad de la microbiota intestinal porque son capaces de inducir respuestas inmunitaria innatas (Green y col. 2011) cuando se detectan microorganismos infecciosos y daño celular. Las mitocondrias también influyen en las actividades de las células efectoras funcionales intestinales, tales como células inmunitarias, células epiteliales y células de enterocromafina (Cunningham y col. 2016). Por otra parte, estas mismas células, están bajo la influencia de la microbiota intestinal, cuyos efectos sobre las funciones de las mitocondria se vuelven cada vez mas evidentes. Por último, los polimorfismos en los genes mitocondriales ND5 y CYTB o los genes de la región D-Loop del genoma mitocondrial se han asociado con composiciones específicas de microbiota intestinal (Ma J. y col. 2014).
Debido a las grandes demandas fisiológicas y a las adaptaciones necesarias durante el ejercicio de alta intensidad, así como a la importancia creciente que la comunicación microbiota intestinal -mitocondrias tiene para la salud intestinal general del huésped, para la producción de energía, la respuesta inmunitaria, la biogénesis mitocondrial y el equilibrio redox, esta revisión se centrará en la evidencia disponible que respalda la existencia de interacción entre mitocondrias y microbiota, así como en los posibles mecanismos fisiológicos involucrados durante el ejercicio de resistencia.
MÉTODOS
Realizamos una revisión sistemática y una síntesis de las investigaciones cualitativas cumpliendo los requisitos establecidos en los elementos de informe preferidos establecidos en los protocolos para la realización de revisiones sistemáticas y metaanalisis (Shamseer y col. 2015). El protocolo fue registrado a priori con PROSPERO el 8 de febrero de 2017 con el Número de identificación CRD42017056852.
Criterios de Elegibilidad y Estrategia de Búsqueda de Bibliografía
Se realizó una búsqueda sistemática e integral en bases de datos electrónicas, entre las que se incluyeron MEDLINE, Scopus, ClinicalTrials.gov, Registro prospectivo internacional de revisiones sistemáticas PROSPERO, Science Direct, Springer Link y EMBASE a partir de enero de 2017 hasta abril de 2017.
En la búsqueda se utilizaron las siguientes palabras clave: “Mitocondrias”, “Biogénesis mitocondrial” “Fosforilación oxidativa”,”estrés oxidativo”, “microbiota intestinal”,”ácidos grasos de cadena corta”,” metabolitos de la microbiota”,”ejercicio de resistencia”, “Inflamación”, “PCG-1α”, “AMPK” y “SIRT1”. La búsqueda no se restringió al tipo de estudio (es decir, especies, meta-análisis, casos y controles, estudios de cohorte prospectivos, revisiones), tamaño de la muestra, año de publicación, estado de publicación o seguimiento; sin embargo, solo consultamos artículos publicados en inglés y no incluimos ninguna tesis de doctorado. Las bibliografías de las revisiones identificadas y de las publicaciones originales fueron seleccionadas a mano con el fin de encontrar estudios adicionales que pudieran no haber sido incluidos en las búsquedas en las bases de datos. Todos los artículos fueron exportados a la base de datos de referencia Zotero. Debido a la naturaleza de esta revisión, no se realizó ninguna solicitud de aprobación por parte del comité de ética.
Extracción y Síntesis de los Datos
Se obtuvieron las copias completas de las citas que fueron señaladas como potencialmente relevantes y las publicaciones que cumplieron con los criterios de inclusión fueron leídas en detalle y se extrajeron los datos. Un revisor (AC) extrajo información sobre el objetivo del estudio, la población y tamaño de la muestra, diseño experimental y duración del seguimiento, especies, características individuales y cambios en la composición de la microbiota intestinal, el metabolismo energético, la actividad redox y la respuesta inmunitaria y la presencia o ausencia de asociación con la función mitocondrial durante el ejercicio de resistencia. La variable principal fue la interacción entre la microbiota intestinal y las mitocondrias en respuesta al ejercicio de alta intensidad que incluyó aspectos como: la regulación ejercida por la microbiota intestinal sobre mediadores clave en la biogénesis mitocondrial (es decir, AMPK, PCG-1α, SIRT1) así como también el estrés oxidativo y la inflamación intestinal inducidos por el ejercicio. Los detalles fueron verificados por un segundo revisor (NM). Si se comprobaba la elegibilidad, se obtenía el artículo completo.
Se leyeron los artículos y los datos extraídos y los hallazgos fueron organizados según: (i) funciones mitocondriales involucradas en la producción de energía, producción de ROS e inflamación durante el ejercicio de resistencia; (ii) estudios experimentales sobre la posible interacción entre la función mitocondrial y la microbiota intestinal; (iii) estudios experimentales en los que se haya demostrado un posible vínculo entre las funciones mitocondriales y los cambios en el perfil de la microbiota intestinal en respuesta los ejercicios de resistencia; (iv) estudios experimentales o revisiones que hubieran observado una relación entre los roles que tienen las variantes genéticas del genoma mitocondrial en las funciones y en el perfil de la microbiota intestinal.
Síntesis de Datos
La búsqueda realizada en enero de 2017 arrojó la siguiente lista de combinaciones de términos clave (microbiota intestinal y producción de energía = 19; microbiota y estrés oxidativo = 22; mitocondrias y estrés oxidativo = 16; mitocondrias, microbiota, ejercicio de resistencia = 1). Un total de 77 estudios experimentales y 84 revisiones cumplieron con los criterios de inclusión y fueron incluidos en la revisión. La mayoría de los artículos fueron revisiones o estudios controlados aleatorizados. El período de recopilación de datos abarcó desde 1980 hasta 2017, y los datos fueron obtenidos en humanos y en modelos animales (es decir, ratones, ratas, caballos, gatos).
DISCUSIÓN
Comunicación Didireccional entre la Microbiota Intestinal y las Funciones Mitocondriales
Las mitocondrias son organelas dinámicas cuya cantidad y volumen cambian en respuesta a la oxidación celular y a las demandas metabólicas. Aunque el genoma mitocondrial es pequeño, el ADN mitocondrial codifica genes que podrían ser esenciales para la producción de energía, el equilibrio redox y la regulación de la inflamación durante el ejercicio de resistencia. El genoma mitocondrial humano está compuesto por ADN circular de 16,6 kb que codifica 13 péptidos involucrados en la fosforilación oxidativa (OXPHOS), dos ribosomas y 22 ARN de transferencia que son cruciales para la síntesis de proteínas intra-mitocondriales. Las funciones de las mitocondrias están bajo doble control genético ejercido tanto por el genoma mitocondrial como por el genoma nuclear. Se sabe que más de 1500 genes codificados por el genoma nuclear (Stewart y Chinnery, 2015) intervienen en las funciones mitocondriales a través de una compleja regulación de mecanismos de transcripción y traducción de genes y ARN no codificantes (Shock y col. 2011).
Durante el ejercicio de resistencia, coactivadores tales como PGC-1α y los factores de transcripción factor respiratorio nuclear 1 y 2 (NRF1, NFR2) (Wu y col., 1999; Hood et al., 2011), el receptor p43 de la hormona tiroidea tri-yodotironina (T3), proteína de unión al elemento de respuesta de AMP cíclico (CREB), el gen supresor tumoral p53, el transductor de señales y activador de la transcripción 3 (STAT3) y todos los receptores de estrógenos controlan la función mitocondrial (Hood y col. 2011). Entre ellos, se ha informado que el PGC-1α sería el principal regulador de la función mitocondrial y de la respiración en los músculos (Hood y col. 2011), especialmente durante ejercicio de resistencia (Steinberg y col. 2006; Wright et al., 2007; Lira y col. 2010). Se observó que durante el ejercicio el PGC-1α experimenta una regulación por incremento y aumenta la cadena de transporte de electrones mitocondriales y también la cantidad de copias de ADN mitocondrial a través de la activación de la subunidad II de la ciclooxigenasa y la unidad IV COX (COX) (Safdar y col. 2011). Además, PGC-1α interviene en la termogénesis, en el metabolismo de la glucosa y en la capacidad oxidativa en diversos tejidos y puede fosforilarse con la proteína quinasa activada por AMP (AMPK), una enzima que también participa en la biogénesis mitocondrial. La AMPK es activada por citoquinas y ejercicio principalmente en respuesta a cambios en la relación AMP: ATP (Lim y col. 2010) y activa a NRF1 y NRF2 (Lee y Wei, 2005). Además, el regulador silencioso 1 (SIRT1), un sensor de energía sensible al equilibrio redox, también puede afectar la biogénesis mitocondrial por desacetilación de PGC-1α (Lakhan y Kirchgessner, 2010; Radak et al., 2013), e intervenir en el cambio en el tipo de fibra muscular (Huang y col. 2016).
Además de la regulación del genoma nuclear y mitocondrial de las funciones mitocondriales, la información genética codificada en todos los microorganismos adquirida a partir del medio ambiente (colectivamente conocido como el microbioma) también regula las funciones mitocondriales modificando la producción de energía, la producción de ROS, la respuesta inflamatoria y los factores de transcripción implicados en la biogénesis mitocondrial. Por definición, cepas individuales de una especie bacteriana pueden diferir hasta en un 30% en términos de secuencia genética (Zhao 2010). Teniendo en cuenta que los genomas de humanos y ratones difieren en solo un 10%, la diversidad genética y funcional dentro de la misma especie bacteriana puede ser abrumadoramente alta (Zhao, 2010). Además, el aporte realizado por estos microorganismos se vuelve realmente impresionante considerando que solo el 10% de la cantidad total de células en el cuerpo humano consiste en células humanas, mientras que el resto proviene de células bacterianas simbióticas (Zhao, 2010).
El análisis filogénico basado en genes ubicados en el genoma mitocondrial indica que las mitocondrias son de origen bacteriano y que evolucionaron a partir de α-proteobacterias (Gray et al., 2001). Se cree que varios parásitos de géneros como Rickettsia (Andersson y col. 1998), Ehrlichia y Anaplasma serían familiares cercanos eubacterianos de las mitocondrias (Gray y col.1999). Aunque la mayoría de los genes de las α-proteobacterias ancestrales han desaparecido del genoma mitocondrial, existiría un estrecho linaje monofilético entre citocromos empleados por bacterias y por mitocondrias tales como las citocromo oxidasas que indican que la cadena respiratoria aeróbica podría ser de origen bacteriano (Kurland y Andersson, 2000). Otros componentes del proteoma mitocondrial provenientes de α-proteobacterias que fueron transferidos a los núcleos son las ATP sintasa 1 y 3 (atp1 y atp3) (Kurland y Andersson, 2000). Por lo tanto, las mitocondrias tienen un genoma diferente y aseguran la síntesis de adenosina trifosfato (ATP) impulsada por el consumo de oxígeno (mediante fosforilación oxidativa, OXPHOS). Lobet y col. (2015) sugirieron que las mitocondrias son un objetivo de elección para los patógenos bacterianos, ya que no son solo un componente clave del metabolismo central si no que también intervienen en la señalización celular a través de la producción de ROS, en el control de la homeostasis del calcio y en la apoptosis celular. Por otro lado, además de las infecciones bacterianas, en los últimos años ha habido algunos experimentos realizados principalmente en animales destinados a explorar cómo la microbiota intestinal comensal modula las funciones mitocondriales (Ma Y. et al., 2014; Mottawea et al., 2016; Saint-Georges- Chaumet y Edeas, 2016). En general, estos estudios han demostrado que las mitocondrias responden a la microbiota intestinal comensal a través de tres formas principales: (i) regulación de la producción de energía, (ii) alteración del equilibrio redox y (iii) regulación de las reacciones inmunitarias respuesta atenuando la oxidación inducida por TNFα y por la inflamación que produce disfunción mitocondrial. Dada la creencia generalizada de que las mitocondrias son simbiontes derivadas de antiguas α-proteobacterias, la interrelación entre las funciones mitocondriales y la microbiota son de gran interés. (Figura 1).
Por otro lado, las mitocondrias pueden modificar la composición de la microbiota comensal y la colonización y adherencia de patógenos a través de diversos mecanismos: (i) producción de ROS y RONS, (ii) inducción de la secreción de células inmunitarias y células de enterocromafina, (iii) modulación de las funciones intestinales, tales como la función de la barrera intestinal y de una adecuada respuesta inmunitaria en la mucosa, que son todos importantes mecanismos para asegurar el mantenimiento de la capa mucosa y de la biopelícula donde crecen grupos individuales de bacterias, (iv) variantes genéticas mitocondriales y heteroplasmia (Figura 1).
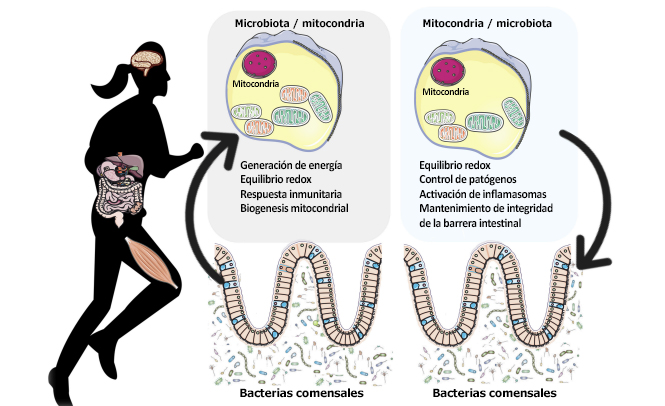
Figura 1. Comunicación bidireccional entre la microbiota intestinal y las mitocondrias. Comunicación desde la microbiota intestinal hacia las mitocondrias: Evidencia reciente demuestra que existe una comunicación bidireccional entre la microbiota intestinal y las mitocondrias. La microbiota y sus subproductos (SCFA y ácidos biliares secundarios) regulan el equilibrio redox y la producción de energía. El metabolismo secundario de los ácidos biliares también podría modificar directamente la biogénesis mitocondrial, la inflamación y la función de la barrera intestinal en diferentes tipos de células (Gao y col. 2009; Korecka et al., 2013; Alex et al., 2014; Caron et al., 2014; Kazgan et al., 2014). En las mitocondrias de los colonocitos, el butirato es sometido a beta oxidación de acidos grasos (FAO) y origina acetil-CoA que entra en el ciclo de los ácidos tricarboxílicos (TCA) y finalmente se produce ATP y CO2 (Donohoe y col. 2011). Entre los ácidos grasos de cadena corta (SCFA), el butirato es un regulador clave de la producción de energía y de la función mitocondrial a través de la expresión del gen PGC-1α en el músculo esquelético y en el tejido adiposo pardo (Gao y col. 2009) y de la mejora en la capacidad respiratoria y de la FAO a través de la activación de la vía AMPK-ACC (Mollica y col. 2017). Comunicación desde las mitocondrias hacia la microbiota: las mitocondrias regulan las funciones intestinales (Igarashi y Guarente, 2016; Wang et al., 2016), tales como la protección de la barrera intestinal (Peng et al. , 2009) y la respuesta inmunitaria de la mucosa, lo que ayuda a mantener la capa de mocosa (Ma Y. y col. 2014) y la microbiota intestinal (Shimada et al., 2012; Caron et al., 2014) SIRT1 mantiene la función de la barrera intestinal a través de varios mecanismos como la mejora de la proliferación de criptas y la supresión de la apoptosis vellosa (Wang y col. 2012), estimulando la expansión de las células madre intestinales en el intestino (Igarashi y Guarente, 2016), regulando la expresión de zonulina ocludina-1, occludina y claudina-1 de las uniones estrechas durante la hipoxia (Ma Y. et al. , 2014). Las variaciones en el genoma mitocondrial pueden afectar la composición de la microbiota intestinal. Por ejemplo, los polimorfismos en los genes ND5 y CYTBo de la región D-Loop del genoma mitocondrial han sido asociados con composiciones específicas de microbiota intestinal como Eubacterium y Roseburia, que son productores de butirato (Ma Y. y col. 2014). Además, el haplotipo europeo HV se ha asociado con una disminución en las probabilidades de sepsis grave, una mayor capacidad de OXPHOS y de producción de ROS y RONS (Jiménez-Sousa y col. 2015) así como también con un elevado VO2máx y una elevada producción de ATP aeróbico en respuesta al ejercicio (Martinez y col. 2010).
Cómo Modula la Microbiota Intestinal las Funciones Mitocondriales
Regulación de la Microbiota Intestinal de la Producción de Energía Mitocondrial
Una de las principales adaptaciones que experimentan los atletas de resistencia en comparación a la población de sujetos no deportistas es la biogénesis mitocondrial y la mejora en el VO2max lo que permite un mayor consumo de oxígeno, y una mejora en la OXPHOS y la FAO del músculo esquelético (Rivera-Brown y Frontera, 2012). El ejercicio de resistencia es el inductor fisiológico de la biogénesis mitocondrial más potente. Se ha observado que el entrenamiento de resistencia regular de 4–6 semanas en humanos y mamíferos aumenta el contenido de mitocondrias de 30 a 100% (Hood y col. 2011) y el volumen hasta un 40% (Hood, 2001; Lundby y Jacobs, 2016). En la misma línea 5 meses de ejercicio de resistencia indujeron biogénesis sistémica mitocondrial, evitaron la degradación y las mutaciones del ADN mitocondrial, incrementaron la capacidad oxidativa mitocondrial y el ensamblaje de la cadena respiratoria, mejoraron la morfología mitocondrial y disminuyeron los niveles patológicos de apoptosis en múltiples tejidos de ratones que presentaban mutaciones en el ADN mitocondrial (Safdar y col. 2011).
La biogénesis mitocondrial se produce por fusión, que es la mezcla de mitocondrias, o por fisión, que es la separación de mitocondrias dañadas (Busquets-Cortés y col. 2016). La mayor cantidad de mitocondrias que se observa en los músculos de atletas entrenados permite un mejor uso de la FAO, de la OXPHOS y del oxígeno, lo que disminuye la oxidación de carbohidratos y, por lo tanto, la degradación de glucógeno y disminuye la producción de lactato (Hood y col. 2011; Rivera-Brown y Frontera, 2012). Por el contrario, una disminución en la cantidad de mitocondrias se relaciona con un deterioro en la capacidad de OXPHOS y de FAO (Wai y Langer, 2016). Más de dos de cada cinco corredores de maratón han expresado que experimentaron un inicio rápido de fatiga e incapacidad para mantener la velocidad y la intensidad del rendimiento debido al agotamiento de carbohidratos (Rapoport, 2010) lo que sugiere que una capacidad deficiente de OXPHOS y de FAO podría ser una causa subyacente importante de la fatiga en atletas (Figura 2).
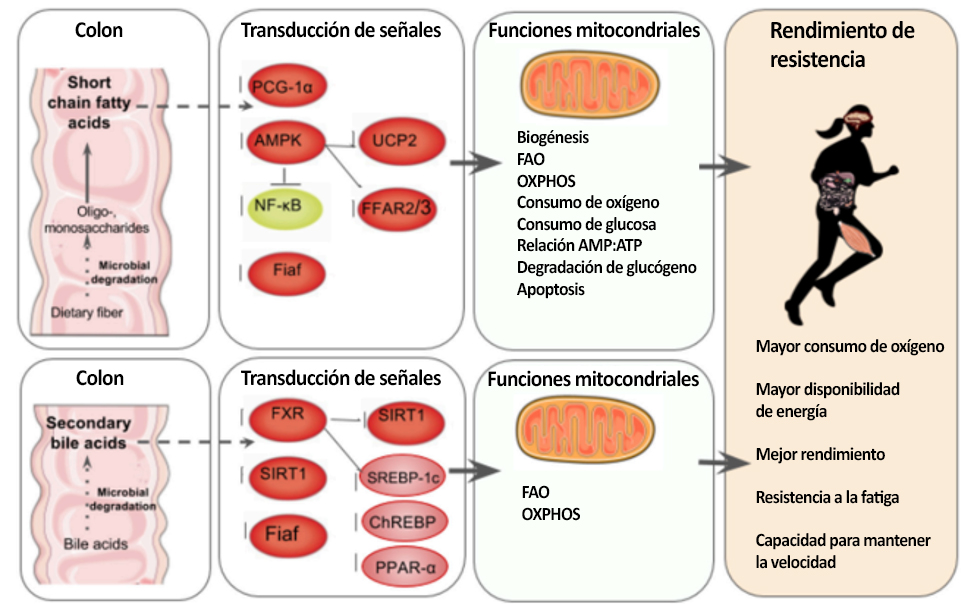
Figura 2. Regulación de la microbiota intestinal de la producción de energía mitocondrial. Arriba, de izquierda a derecha: en el colon, la microbiota intestinal fermenta las fibras dietéticas no digeribles tales como el almidón resistente y los oligosacáridos para producir SCFA en los intestinos, quienes pueden representar hasta el 10% de los requerimientos calóricos humanos (den Besten et al. 2013). Los SCFA son mediadores clave del metabolismo energético de las mitocondrias y actúan como ligandos para los receptores de ácidos grasos libres 2 y 3 (FFAR2, FFAR3) que regulan el metabolismo de la glucosa y de los ácidos grasos (den Besten y col. 2013; Kimura et al., 2014). Los SCFA regulan a SIRT1 que interviene en la biogénesis mitocondrial a través de la desacetilación de PGC-1α (Lakhan y Kirchgessner, 2010; Radak et al., 2013). En las células del músculo esquelético, el butirato fosforila al AMPK y al p38, quienes luego activan PGC-1α y, por lo tanto, la producción de la FAO y de ATP. El butirato también activa AMPK a través de la vía UCP2-AMPK-ACC (den Besten y col. 2015). Las bacterias comensales tales como Lactobacillus rhamnosus CNCMI – 4317 se ha asociado con una mayor expresión de Fiaf (Jacouton y col. 2015). En los macrófagos de la lámina propia, los SCFA también inhiben la activación de NF-κB lo que reduce la inflamación asociada con la colitis ulcerosa (Lührs y col. 2002). El resultado es un aumento en la biogénesis mitocondrial, la FAO, la OXPHOS, el consumo de oxígeno, la absorción de glucosa, la relación de AMP:ATP y la degradación de glucógeno y una menor apoptosis (Lantier et al ., 2014; Canfora et al., 2015; den Besten et al., 2015). Abajo, de izquierda a derecha: las bacterias anaerobicas degradan del 5 al 10% de los ácidos biliares (Gérard, 2013), y los ácidos biliares secundarios regulan el metabolismo de carbohidratos y lípidos a través de la modulación de los receptores del factor de transcripción del receptor farnesoide X (FXR) y de la proteína de membrana 5 acoplada a G (TGR5) lo que produce un incremento en la beta-oxidación de ácidos grasos (FAO) y en la fosforilación oxidativa (OXPHOS) ( Nie et al. 2015). FXR regula el metabolismo de los carbohidratos mediante la regulación de la expresión de SIRT1 y Fiaf así como por la activación de SREBP-1c y ChREBP ( Kuipers et al. , 2014; Joyce y Gahn, 2016) y el metabolismo de los ácidos grasos a través de la activación de PPAR-α (Joyce y Gahn, 2016). Cada vez existe mayor cantidad de evidencia que demuestra que el metabolismo secundario de los ácidos biliares también podría modificar directamente la biogénesis mitocondrial, la inflamación y la función de barrera intestinal en diferentes tipos de células (Gao y col. 2009; Korecka et al., 2013; Alex et al. 2014 ; Caron et al., 2014; Kazgan et al., 2014). El efecto de la actividad de los SCFA y de los ácidos biliares secundarios en la biogénesis mitocondrial es un mejor rendimiento deportivo general debido a un mejor consumo de oxígeno, a una mayor disponibilidad de energía y a una mayor resistencia a la fatiga.
Debido a los requerimientos de energía durante el ejercicio de resistencia, y a la relación recíproca y compleja descripta recientemente entre la microbiota intestinal y el metabolismo energético de todo el cuerpo, no es sorprendente que se hayan incrementado los esfuerzos para identificar los mecanismos por los cuales la microbiota intestinal mejora la FAO y la OXPHOS mitocondrial en los deportistas de élite (Pyne y col. 2015).
La microbiota intestinal contiene más de 100 billones de microorganismos (Rajilic-Stojanovic y de Vos , 2014), distribuidos aproximadamente en 160 especies y contienen 9 millones de genes (Li et al., 2014) que son clave para el metabolismo energético del huésped. En el intestino, las bacterias anaerobicas fermentan y extraen energía de polisacáridos no digeribles, como las fibras y el almidón resistente, y sintetizan SCFA como el N-butirato, propionato y acetato (Flint y col. 2008). Dietas altas en fibra permiten producción de 400–600 mmol de SCFA en el ciego por día, lo que representa aproximadamente el 10% de los requerimientos calóricos humanos (den Besten et al., 2013). En los colonocitos, el butirato es transportado hacia las mitocondrias donde es metabolizado por betaoxidación de ácidos grasos (FAO) en condiciones aeróbicas y se convierte en acetil-CoA, que ingresa al ciclo de Krebs y da como producto NADH que luego ingresa en la cadena de transporte de electrones y finalmente produce ATP y CO2 (Donohoe y col. 2011). Además, el butirato es oxidado en las mitocondrias por una serie de cinco enzimas, entre las que se incluye la enzima mitocondrial acetoacetil CoA tiolasa y se ha observado que la oxidación de butirato está afectada en pacientes con colitis ulcerosa (Roediger y col. 1993; Ahmad et al., 2000; Santhanam et al., 2007) lo que sugiere que el butirato desempeña un papel importante en la actividad del ciclo de los ácidos tricarboxilicos (TCA) en colonocitos y, por lo tanto, en la salud general del colon. Además, el propionato y el acetato pueden ser transportados al torrente sanguíneo hacia diferentes órganos donde son utilizados como sustratos para la oxidación mitocondrial, producción de lípidos y gluconeogénesis, que se sintetiza a partir de propionato (Nicholson y col. 2012).
Aunque faltan estudios en humanos, varios estudios realizados con animales libres de gérmenes (GF) o sin patógenos específicos (SPF) han demostrado que los SCFA, como el N-butirato y el acetato, pueden afectar el metabolismo energético mitocondrial a través de una amplia gama de factores de transcripción que controlan directa o indirectamente las funciones mitocondriales. Los tipos y la cantidad de SCFA producidos por microorganismos intestinales dependen de la composición de la microbiota intestinal, de las interacciones metabólicas entre las especies de microorganismos y de la cantidad y tipo de los principales macro y micronutrientes de la dieta (den Besten y col. 2013). Mientras mas polisacáridos de origen vegetal, oligosacáridos, almidón resistente y fibra dietética se consuma, más fermentarán estas bacterias estas fuentes de alimentos no digeribles en SCFA beneficiosos.
Los estudios realizados con ratones C57BL/ 6 han demostrado que una inyección de butirato de sodio 5% p/p además de una dieta alta en grasas (58% de las calorías provenientes de la grasa) logró prevenir la resistencia a la insulina al estimular la termogénesis y la oxidación de ácidos grasos en las mitocondrias del músculo esquelético y del tejido adiposo pardo, en parte debido a una mayor expresión del gen PGC-1α y a una mayor actividad proteica (Gao y col. 2009). Notablemente, la inducción de PGC-1α mediada por butirato produjo una transformación de fibras musculares esqueléticas de tipo II (glucolítico) a tipo I (oxidativo), que son ricas en mitocondrias y estimulan la FAO para la producción de ATP (Gao y col. 2009). De manera similar, ratas alimentadas con leche humana en comparación con leche de vaca o de burro presentaron una mayor eficiencia energética de las mitocondrias asociada con cambios en la capacidad de la microbiota para producir N-butirato (Trinchese y col. 2015). Por lo tanto, es interesante especular que el N-butirato podría desempeñar un papel importante en la activación de PGC-1α, que es un marcador biológico de funciones mitocondriales durante la resistencia debido a que se ha observado que la expresión del gen PGC-1αaumenta marcadamente en el músculo esquelético en respuesta al ejercicio (Pilegaard y col. 2003; Bo et al. 2010; Little et al., 2010; Safdar et al., 2011).
El N-butirato y el acetato también pueden afectar la función y el metabolismo mitocondrial a través de la activación de AMPK en los colonocitos (Canfora et al. 2015 ; den Besten et al., 2015). Se ha demostrado que el AMPK es un sensor de energía clave en el músculo esquelético y que es capaz de regular la OXPHOS mitocondrial (Lantier y col. 2014 ). den Besten y col. (2015) descubrieron que SCFA activaba la vía de la UCP2-AMPK-acetil CoA carboxilasa (ACC) que regula hacia la baja la expresión génica de PPARγ lo que produce una disminución de la lipogénesis y un aumento en la relación AMP:ATP. El aumento de la relación AMP: ATP también puede activar a AMPK en hígado, tejido adiposo (Richter y Ruderman, 2009) y en el musculo (Hood y col. 2011), lo que estimula el consumo de glucosa, la FAO y la OXPHOS mitocondriales y disminuye la síntesis de proteínas y de lípidos. Del mismo modo, Mollica et al. (2017) observaron que el N-butirato mejoró la capacidad respiratoria y la FAO a través de la activación de la vía AMPK-ACC, lo que promovió fusión mitocondrial en el hígado y reducción de la resistencia a la insulina y acumulación de grasa en ratones obesos. Los SCFA también son mediadores clave del metabolismo energético de las mitocondrias porque sirven como ligando para los receptores de ácidos grasos libres 2 y 3 (FFAR2, FFAR3), también conocidos como receptores acoplados a proteínas G (GRP43) y GRP41 respectivamente, que regulan el metabolismo de la glucosa y los ácidos grasos (revisado extensamente por den Besten et al., 2013; Kimura et al., 2014), así como GPR109A que activa las vías asociadas con la homeostasis energética, el almacenamiento de lípidos y la ingesta de alimentos (Nicholson et al. 2012).
El impacto de la microbiota en las funciones mitocondriales ha sido respaldado por estudios cuya finalidad consiste en manipular la microbiota intestinal mediante el uso de probióticos. La administración del probiótico Lactobacillus rhamnosus CNCMI – 4317 se asoció con una mayor expresión de Fiaf también conocida como ANGTPL4 (angiopoyetina 4), a través de vías dependientes de PPAR-α (Jacouton y col. 2015) lo que modificó la capacidad de realizar OXPHOS de las mitocondrias. En línea con esto, Bäckhed et al. (2007) demostraron que los ratones GF expresaban un fenotipo magro porque estaban protegidos de los efectos obesogénicos de una dieta occidental rica en grasas y azúcares gracias a la elevada expresión de Fiaf en los intestinos, así como por la mayor expresión de AMPK y PGC-1α en el músculo esquelético y en el hígado, quienes regulan la OXPHOS de las mitocondrias en las células musculares (Lantier y col. 2014). Finalmente ciertas bacterias intestinales como Eubacterium hallii y Anaerostipes caccae tienen la capacidad de transformar el subproducto de la glucólisis anaeróbica, el lactato en SCFA cuando se ha agotado la glucosa generando así una fuente de energía alternativa para el huésped (Duncan y col. 2004; Scott et al., 2013) que no depende de la OXPHOS (Rogatzki et al., 2015).
Además de SCFA, los ácidos biliares secundarios producidos por la microbiota intestinal también desempeñan un papel importante en la regulación del metabolismo de la energía mitocondrial. Las bacterias anaeróbicas de los géneros Bacteroides, Eubacterium y Clostridium degradan 5–10% de los ácidos biliares primarios que forman ácidos biliares secundarios (Gérard, 2013). Los ácidos biliares secundarios interactúan con las mitocondrias modulando los factores de transcripción relacionados con el metabolismo de lípidos y de carbohidratos, entre los que se incluyen el receptor farnesoide X (FXR) y la proteína de membrana acoplada a G 5 (TGR5) (Nie y col. 2015). El FXR es un objetivo de la proteína desacetilasa dependiente de NAD SIRT1 (revisado por Kuipers et al. , 2014) y regula a la proteína de unión al elemento regulador del esterol (SREBP-1c), a la proteína de unión al elemento sensible a los carbohidratos (ChREBP) y PPAR-α, que estimula la absorción y oxidación de ácidos grasos (Joyce y Gahn 2016).
Cada vez existe mayor cantidad de evidencia que el metabolismo de los acidos biliares secundarios puede modificar directamente la expresión de SIRT1 y de Fiaf, así como también la biogénesis mitocondrial, la inflamación y el funcionamiento de la barrera intestinal en diferentes tipos de células (Gao et al., 2009; Korecka y col. 2013; Alex et al., 2014; Caron et al., 2014; Kazgan y col. 2014). También se ha comenzado a estudiar el rol de SIRT1 en la homeostasis intestinal lo que arrojará claridad sobre cual es el rol que la comunicación microbiota intestinal-mitocondria tiene en el metabolismo energético (Kazgan y col. 2014; Lo Sasso et al., 2014). Debido a que un ejercicio en rueda de 12 semanas realizado por ratones de tipo salvaje mejoró la secreción biliar de ácidos biliares y aumentó la eliminación fecal de ácido biliar y de esteroles en comparación con el grupo control de ratones sedentarios (Meissner y col. 2011), es tentador especular sobre el papel que desempeña la microbiota en metabolismo energético a través de la regulación de los ácidos biliares, los SCFA y de la inducción indirecta de genes SIRT1, Fiaf y FXR (Figura 2).
A diferencia de los efectos beneficiosos que tienen las bacterias comensales en el metabolismo energético, patógenos como Salmonella y Escherichia coli (Leschelle y col. 2005) puede producir efectos negativos para el metabolismo energético de las mitocondrias del huésped a causa de la degradación de aminoácidos azufrados y la producción de sulfuro de hidrógeno (H2S) en el intestino grueso. El H2S es un mediador importante de muchos procesos fisiológicos y patológicos. Altas cantidades de H2S pueden inhibir un componente clave de la cadena respiratoria mitocondrial al penetrar las membranas celulares e inhibir la actividad COX y la producción de energía (Blachier y col. 2007; Mottawea y col. 2016). Los patobiontes también pueden producir NO, lo que puede afectar la actividad mitocondrial del huésped y favorecer la infección bacteriana (Vermeiren et al., 2012). Además de los patobiontes, las dietas altas en proteínas, que son comunes en muchos atletas de élite, pueden dar lugar a altos niveles de H2S y de urea en el intestino (revisado por Windey et al. , 2012). Por ejemplo, en humanos es posible alcanzar concentraciones fecales de H2S de hasta 3,4 mM cuando se consume una dieta alta en proteínas (Leschelle et al., 2005). Las altas concentraciones del grupo con H2S intestinal provocaron una disminución en la expresión de COX en el las células HT-29 de colon humano y, por lo tanto, una reducción en la transferencia de electrones de los complejos I y II en la cadena respiratoria produciendo un cambio desde metabolismo oxidativo hacia la glucólisis, aumento de la producción de lactato y disminución de la producción de ATP (Leschelle y col. 2005). Del mismo modo, Beaumont et al. (2016) concluyeron que la exposición de las células humanas HT-29 a elevados niveles de H2S no solo provocó un menor consumo de oxígeno mitocondrial si no que también un aumento en la expresión de genes inflamatorios tales como IL-6, que se incrementaban después de una dieta alta en proteínas. Mottawea y col. (2016) demostraron recientemente que la proliferación de patobiontes, muchos de los cuales son potentes productores de H2S, producen una regulación hacia la baja de las proteínas mitocondriales.
Además, el H2S puede inhibir la β-oxidación de butirato en el colon, lo que se cree que podría producir colitis ulcerosa (Leschelle y col. 2005; Blachier et al., 2007). En línea con esto, Le Chatelier et al. (2013) estudiaron la diversidad microbiana en personas obesas versus personas sanas. Descubrieron que los individuos que tenían una gran diversidad bacteriana tenían una mayor producción de hidrógeno y de ácidos orgánicos (es decir, N-butirato, propionato) mientras que aquellos que tenían una baja diversidad bacteriana tenían menos bacterias productoras de N-butirato, un mayor potencial de liberación de H2S y abundancia de Campylobacter / Shigella y de microbiota asociada a inflamación general.
Dada la falta de estudios en atletas de resistencia, es difícil hacer recomendaciones dietéticas precisas para los atletas de resistencia en cuanto a cómo optimizar el metabolismo de SCFA y de ácidos biliares para la producción de energía. Sin embargo, el consumo de una gran cantidad de proteínas animales durante los días de descanso y el entrenamiento puede ser negativo y podría afectar la microbiota intestinal de atletas de élite (p. ej., producción de subproductos potencialmente tóxicos como H2S). Está claro entonces que la interacción entre dieta y ejercicio debe ser estudiada más a fondo para evaluar mejor las contribuciones de la dieta y de las actividades microbianas en las funciones de las mitocondrias y en el rendimiento deportivo.
Regulación de la Microbiota Intestinal de la Producción de ROS Mitocondrial
Durante y después del ejercicio, aumenta la producción de ROS y RONS en respuesta a las necesidades energéticas y se producen principalmente a partir de la pérdida de electrones en la cadena de transporte de electrones mitocondriales (Radak y col. 2013). El complejo I de la cadena de transporte de electrones mitocondrial es uno de los mayores generadores de ROS, RONS, y radicales libres como NO y anión superóxido (O-2 ), que son moléculas que son inestables porque poseen una alteración electrónica que causa reacciones de oxidación con proteínas, lípidos y ADN para convertirse en moléculas estables (Fisher-Wellman y Bloomer, 2009; Hood et al., 2011; Gomes et al., 2012; Radak y col. 2013).
La producción de ROS inducida por el ejercicio durante la actividad física regular puede producir adaptaciones beneficiosas al ejercicio, como vasoregulación, proliferación de fibroblastos (Gomes y col. 2012), hipertrofia muscular, aumento de la biogénesis mitocondrial, inducción de enzimas antioxidantes (Radak y col. 2013) y regulación de la respuesta inmunitaria para eliminar antígenos ( Fisher-Wellman y Bloomer, 2009; Radak et al., 2013). Diez días del ejercicio de resistencia pueden elevar la producción de antioxidantes, disminuir la inflamación y reducir la permeabilidad intestinal (Holland y col. 2015). La hipótesis que se ha aceptado generalmente es que las ROS mitocondriales pueden inducir NF-κβ y a la proteína activadora 1 (AP-1) que activa enzimas antioxidantes como la manganeso superóxido dismutasa (Mn-SOD) (Radak y col. 2013), catalasa y SOD (Blaser y col. 2016), y también a PGC-1α (Steinberg et al., 2006), por lo que tendrian un efecto homeostático (Blaser et al. , 2016). En ratones knockout para PGC-1α, St-Pierre et al. (2006) observaron un disminución en los niveles de Mn-SOD y de superóxido dismutasa de cobre-zinc, lo que sugiere que los ratones knockout PGC-1α son más sensibles al estrés oxidativo (St-Pierre y col. 2006).
Por otra parte, muchos atletas sufren de estrés y entran en un círculo vicioso de sobreesfuerzo por el entrenamiento extenuante y las competencias, y pueden presentar niveles crónicamente altos de ROS y RONS que pueden agotar el sistema antioxidante no enzimático y dañar el funcionamiento celular. En atletas sobre entrenados, la liberación excesiva de hormonas del estrés inducida por el esfuerzo físico y el mayor consumo de oxígeno en el cuerpo, podrían conducir a la generación de ROS y RONS en los tejidos que sufren isquemia e hipoperfusión (Mach y col. 2017). La hiperpermeabilidad intestinal inducida por la isquemia se produce generalmente en sujetos que hacen ejercicio al 70% consumo de oxígeno máximo cuando se reduce el suministro de sangre en al menos un 50% (Holland y col. 2015). Por lo tanto, los atletas tienen dos fuentes principales de ROS y RONS: la cadena de transporte de electrones en las mitocondrias y los intestinos a través de las células epiteliales y los neutrófilos transmigrantes en la luz intestinal (Figura 3).
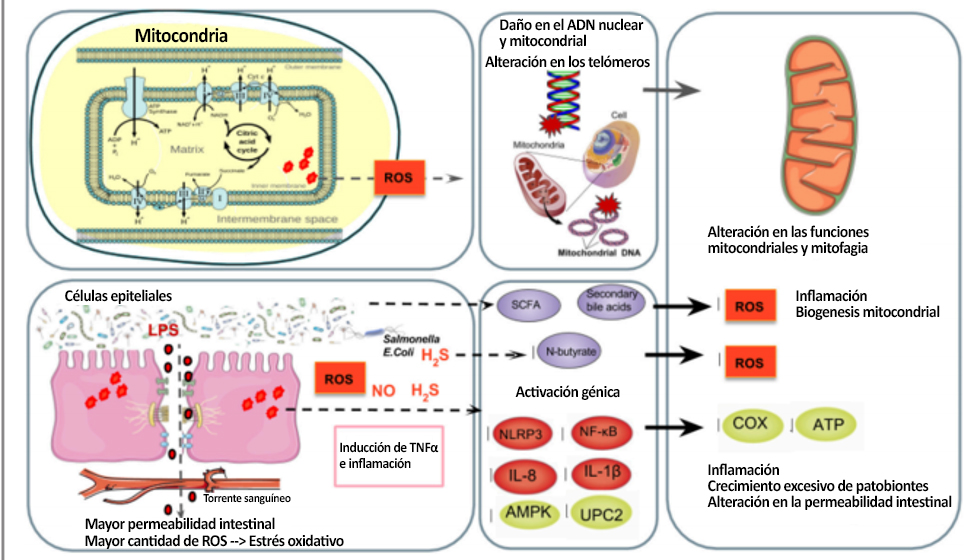
Figura 3. Regulación de la microbiota intestinal de la producción de ROS mitocondriales. Arriba, de izquierda a derecha: los atletas tienen dos fuentes principales de ROS y RONS: la cadena de transporte de electrones mitocondriales y las células epiteliales intestinales y neutrófilos transmigrantes en la luz intestinal (Holland y col. 2015) en donde se producen radicales libres tales como NO y superóxido (Fisher-Wellman y Bloomer, 2009; Gomes et al., 2012; Radak et al., 2013). Individuos poco entrenados y atletas sobreentrenados tienen un mayor riesgo de sufrir estrés oxidativo (Radak y col. 2008) que provoca daño en el ADN inducido por ROS (Radak et al. , 2013), aumento en las mutaciones del ADN (Green et al. , 2011), acortamiento de la longitud de los telómeros (Wallace et al. , 2010) y alteración en la biogénesis mitocondrial (Sahin et al., 2011). Abajo de izquierda a derecha: la excesiva liberación de hormonas de estrés que experimentan los atletas sobreentrenados, así como el aumento en el consumo de oxígeno en el cuerpo pueden generar ROS y RONS en los tejidos que sufren isquemia e hipoperfusión (Mach y col. 2017). La hiperpermeabilidad intestinal inducida por isquemia (Holland et al., 2015) puede inducir la translocación de LPS y una cascada inflamatoria de TNFα (Clark y Mach , 2016), la inhibición de OXPHOS desencadenada por ROS y el inflamasoma NLRP3 los que provoca una respuesta inflamatoria mediada por mitocondrias (Green y col. 2011; de Zoete y Flavell, 2013) y mitofagia (Shimada et al. , 2012), así como la expresión de NF-κB, IL-1β, IL-6 e IL-8 (Liu et al. 2012). TNFα e IL-6 inhiben la activación de AMPK, lo que reduce el metabolismo de la glucosa y la FAO en las mitocondrias (Steinberg et al., 2006; Lim et al., 2010; Viollet et al. 2010; Andreasen et al., 2011). La expresión reducida de la proteína desacoplada 2 (UPC2) puede provocar el desacoplamiento parcial de la fosforilación oxidativa (OXPHOS) mitocondrial (Crouser et al., 2002) y una producción elevada de ROS (Saint-Georges-Chaumet et al. , 2015). Además, los patobiontes (es decir, Fusobacterium, Veillonella y Atopobium parvulum) pueden producir sulfuro de hidrógeno (H2S) y óxido nítrico (NO) lo que favorece la proliferación de infecciones y la inflamación (Mottawea y col. 2016), inhibición de la actividad COX y de β-oxidación de butirato en el colon (Leschelle y col. 2005; Blachier et al., 2007) lo que afecta negativamente el funcionamiento y la producción de energía mitocondrial (Blachier et al., 2007; Mottawea et al., 2016). Por otro lado, los SCFA como el N-butirato y los ácidos biliares secundarios pueden influir en las funciones mitocondriales relacionadas con la producción de energía, la biogénesis mitocondrial, el equilibrio redox y las cascadas inflamatorias, lo que lo convierte en un objetivo terapéutico potencial para la resistencia (Circu y Aw, 2012; Bar et al. 2013 ; den Besten et al., 2013; Mottawea et al., 2016) .
Parece que tanto los individuos poco entrenados como los atletas sobreentrenados tienen un mayor riesgo de sufrir estrés oxidativo (Radak y col. 2008), que se caracteriza por un aumento sustancial en el daño por ROS en lípidos, proteínas y ADN (Hood et al. 2011; Radak et al., 2013). El daño oxidativo del ADN aumenta la tasa de mutaciones en el genoma mitocondrial y nuclear (Green y col. 2011). Es importante destacar que el ADN mitocondrial es más susceptible a daño oxidativo y a la acumulación de mutaciones debido a la cercanía con las especies reactivas de oxigeno (ROS) que se producen en las mitocondrias (Lee y Wei, 2005; Crane et al., 2013). Por otra parte, estudios recientes han revelado que la producción de ROS también reduce la longitud de los telómeros y modifica la biogénesis mitocondrial (Sahin y col. 2011). La disfunción de los telómeros activa la vía mediada por p53 para reprimir la expresión de PPAR-γ, PGC-1α y PGC-1 (Sahin y DePinho, 2012). A su vez, la represión de ambos coactivadores deteriora la biogénesis mitocondrial funcional lo que provoca mayores niveles de ROS que dañan tanto los telómeros como el ADN mitocondrial, y se inicia un ciclo de retroalimentación negativa. Además, varios estudios informaron recientemente que se han observado correlaciones positivas entre la longitud de los telómeros y la variación en la cantidad de copias de ADN mitocondrial en adultos sanos (Kim y col. 2013) y entre la longitud de los telómeros y el envejecimiento y varias enfermedades (Garatachea y col. 2015).
Palazzetti y col. (2003) analizaron los niveles de estrés oxidativo en varones triatletas versus individuos sedentarios antes y después de un período de sobre entrenamiento de 4 semanas a través de muestras de sangre y orina. Los autores observaron que el sobre entrenamiento podía comprometer el mecanismo de defensa antioxidante debido a un aumento en la peroxidación de lípidos y a la regulación por incremento de la glutatión peroxidasa plasmática (GSH-Px), que no lograba evitar el daño oxidativo, lo que coincide con lo observado en otros estudios realizados con atletas sobreentrenados (Palezzetti et al., 2003). Bloomer y col. (2005) también demostraron que solo 30 min de ejercicio anaeróbico y aeróbico agudos también pueden provocar desequilibrio redox tal como se evidencia por el aumento significativo en la oxidación de glutatión (disminución de GSH y aumento de GSSG después del ejercicio), un aumento de la peroxidación de lípidos (proteínas carbonilos) y un aumento menor en la oxidación del ADN (8-OHdG). En un estudio realizado en ratones, Li y col. (2015) observaron que el ejercicio agudo (76% VO2máx. durante 15, 60 o 90 min) indujo estrés oxidativo mitocondrial. Esto fue confirmado por una mayor producción de ROS, quienes tienen la capacidad de inducir a la familia de receptores de tipo NOD inflamatorios y a NLRP3. Del mismo modo, se demostró recientemente en caballos, quienes son considerados como un modelo óptimo in vivo para caracterizar la respuesta al ejercicio de resistencia, que las tasas elevadas de respiración durante el ejercicio de resistencia habían desencadenado la generación de una cantidad de ROS mayor a la que los sistemas antioxidantes podían neutralizar (Mach et al., 2016, 2017). Es importante destacar que muchos estudios realizados con atletas obtienen resultados variables en los marcadores oxidativos debido a la heterogeneidad de la metodología, al tipo de análisis de sangre, de orina y de ADN que emplean, al estado de entrenamiento del atleta, tipo de ejercicio, condiciones de ejercicio, a los efectos a largo plazo del sobreentrenamiento y a la dieta de atleta. Por lo tanto, es difícil hacer generalizaciones sobre el estado oxidativo pre y post ejercicio en atletas de resistencia; sin embargo, parece que en general, los atletas bien entrenados que entrenan 20–30 h por semana probablemente tienen una menor producción de RON y mejores mecanismos de defensa antioxidante y por lo tanto, un mejor balance redox (revisión de Fisher-Wellman y Bloomer, 2009).
En los últimos años se observó una proliferación de trabajos experimentales realizados principalmente en animales, destinados a explorar cómo la microbiota modula la producción mitocondrial de ROS. Como se mencionó anteriormente, la evidencia indica que la microbiota intestinal se comunica con las mitocondrias a través de los SCFA, que tienen propiedades antioxidantes. La ausencia de colonización microbiana en ratones se asoció con niveles reducidos de SCFA y niveles séricos inferiores de GPx y catalasa después de ejercicios de natación de resistencia, que son antioxidantes indispensables para reducir el estrés oxidativo (Hsu y col. 2015). Diferentes estudios realizados con modelos de ratones (Dobashi et al., 2011, 2013) han informado que la microbiota intestinal regula la actividad de la superóxido dismutasa (SOD), pero se necesitan más estudios para comprender mejor el papel de la microbiota intestinal en el control del equilibrio redox intestinal en respuesta al ejercicio de alta intensidad a largo plazo, la inflamación y la disbiosis intestinal.
Otros estudios han demostrado que los SCFA puede inhibir el acortamiento de la longitud de los telómeros (Garatachea y col. 2015). O’Callaghan y col. (2012) descubrieron que una mayor producción de SCFA en el colon en ratas se asociaba con niveles reducidos de malondialdehído (un marcador de estrés oxidativo), acortamiento de los telómeros y daño en el ADN. Aunque no se conocen los mecanismos exactos de cómo los SCFA redujeron el daño de los telómeros en este experimento, otros estudios han demostrado que el N-butirato posee propiedades antioxidantes que pueden reducir el daño inducido por H2O-2en el ADN posiblemente por alteración de las estructuras de la cromatina como la longitud de los telómeros y pueden inducir la producción del antioxidante glutatión (Rosignoli et al ., 2001; Hamer et al., 2009). El N-butirato puede reducir los niveles de H2O2 en el colon y, por lo tanto, disminuir daño oxidativo al aumentar la actividad de COX-2 (Martinez et al. 2010). En resumen, los SCFA pueden mejorar la función mitocondrial reduciendo los niveles de ROS a través de varios mecanismos posibles que pueden afectar el ADN y acortar los telómeros, dos factores asociados con el envejecimiento y la alteración en la función mitocondrial.
La microbiota intestinal también puede metabolizar el aminoácido aromático triptófano, que además de ser el precursor del neurotransmisor serotonina también desempeña un papel en la síntesis de cofactores para reacciones redox, nicotinamida adenina dinucleótido (NAD) y NAD fosfato (NADP) (Ito et al., 2003 ; Richard et al., 2009). Otras bacterias podrían usar triptófano para producir ácido quinolínico, que es un metabolito neuroactivo de la ruta de la kinurenina a menudo implicado en la patogenia de una variedad de enfermedades neurológicas humanas y peroxidación lipídica (Kurnasov y col. 2003). Un análisis genómico comparativo demostró que varias bacterias contienen enzimas bacterianas como la 3-hidroxi-kinureninasa, necesaria para convertir el triptófano en ácido quinolínico (Kurnasov et al., 2003). Strephtomyces antibioticus, Cyanidium caldarium, Karlingia rosea y Xanthomonas pruni usan triptófano para sintetizar ácido quinolínico y Pseudomonas aureofaciens posee enzimas como la triptófano dioxigenasa y la quinureninasa (Kurnasov y col. 2003). Otros patobiontes como Bacteroides thetaiotaomicron, Proteus vulgaris y E. coli poseen la enzima triptofanasa bacteriana, que transforma el triptófano en metabolitos como indol-3-piruvato, indol-3-lactato, y indol-3-acetato (Boulangé y col. 2016). El indol-3-piruvato puede convertirse corriente abajo en indol-3-acetato, que tiene la capacidad de activar los peróxidos de rábano picante causando la formación de radicales libres y peroxidación lipídica (Kumavath et al., 2017). Por último, algunos patobiontes intestinales han adaptado sus respuestas reguladoras frente al estrés oxidativo (Chiang y Schellhorn, 2012) que pueden favorecer el crecimiento de patógenos o inhibir la producción de N-butirato (Rivera-Chávez y cols. 2017). Por ejemplo, el aumento del contenido de nitrato en el colon favorece la proliferación de enterobacterias patógenas como E. coli y Salmonella spp. a través de la respiración de nitrato (Rivera-Chávez et al., 2017; Zeng et al., 2017). Estos resultados demuestran que las bacterias no comensales podrían producir desbalances redox e inducir las vías inflamatorias en el intestino.
Ghosh y col. (2011) afirman que la producción de ROS es un mecanismo de defensa que puede provocar citotoxicidad contra los patógenos y reducir la carga de infección en el hospedador. De hecho, Ghosh et al. (2011) demostraron que la infección por Citrobacter rodentium en C57BL/6 resistente dio como resultado más Bacteroides y niveles elevados de estrés oxidativo (menor relación GSH, inducción de iNOS y una menor expresión de proteínas antioxidantes MnSOD/SOD2). Una revisión de Lobet et al.(2015) describe cómo la producción de ROS ha estado involucrada en la eliminación de diferentes patógenos intracelulares como la listeria monocytogenes, Staphilococcus typhimurium o Toxoplasma gondii. Sin embargo, se ha demostrado que para superar el efecto mitocondrial sobre la respuesta inmune y la supervivencia celular, numerosas especies bacterianas de microbiota tienden a reducir directamente la producción de ROS mitocondriales (Lobet y col. 2015). Por ejemplo, Mycobacterium tuberculosis regula a la baja la vías de señalización TLR inducidas por LPS lo que reduce la producción de ROS mitocondriales (Saint-Georges-Chaumet y Edeas, 2016). Otras toxinas microbianas pueden aumentar la actividad de la enzima de desintoxicación superóxido dismutasa mitocondrial, lo que provoca una disminución del contenido de ROS y reduce la apoptosis en las células del huésped, tal como se observa en Ehrlichia chaffeensis (Liu y col. 2012).
Regulación de la Actividad Inflamatoria Mitocondrial por parte de la Microbiota Intestinal
Según una revisión de Mach y Fuster-Botella (en prensa), el ejercicio intenso aumenta los niveles plasmáticos de cortisol induciendo la afluencia de neutrófilos desde la médula ósea y el flujo de salida de otros subtipos de leucocitos, lo que provoca una respuesta inflamatoria de fase aguda. A diferencia con lo que ocurre en el ejercicio liviano habitual y en el fitness (Clarke y col. 2014), el ejercicio extenuante provoca un aumento en la cantidad de citoquinas proinflamatorias, tales como TNFα, IL-1, IL- 6, del antagonista del receptor IL-1, de los receptores de TNF, así como de los moduladores antiinflamatorios tales como IL-10, IL-8 y proteína-1 inflamatoria de macrófagos, lo que indica que existiría un efecto dosis-respuesta entre las respuestas biológicas al ejercicio y la inmunidad del huésped (revisado por Clark y Mach, 2016).
Un mecanismo clave de la inflamación es la activación de inflamasomas, que son grandes complejos de proteínas citosólicas que activan la caspasa-1, que induce las citocinas inflamatorias IL- 1 e IL-18 y estimulan a otros mediadores inflamatorios que son cruciales para la respuesta inmunitaria innata. Las mitocondrias desempeñan un papel central en el inicio de inflamasomas y de otras vías inflamatorias y como tal participan en la autofagia, la muerte celular y la inflamación (Green y col. 2011). Aunque actualmente se desconocen los mecanismos exactos, los agentes no infecciosos tales como los inhibidores de la fosforilación oxidativa liberadores de ROS y las señales proinflamatorias parecen activar NLRP3, lo que desencadena una respuesta inflamatoria mediada por mitocondrias (Green y col. 2011; de Zoete y Flavell, 2013). La activación de NLRP3 también promueve mitofagia (autofagia mitocondrial), un proceso celular que elimina las mitocondrias cuyo funcionamiento esta alterado (Shimada y col. 2012). Como se mencionó anteriormente, Li et al. (2015) informaron que los ratones que realizaron ejercicio agudo (76% VO2 máx. durante 15, 60 o 90 min) experimentaron estrés mitocondrial inducido por el ejercicio, que fue confirmado por una mayor producción de ROS y por la mayor expresión de NLRP3.
Otra forma en que el entrenamiento de resistencia puede provocar cambios en la respuesta inmune y en la función mitocondrial es a traves de la reducción del flujo sanguíneo gastrointestinal, de oxígeno y de nutrientes mientras aumenta la hipertermia tisular, la permeabilidad de la pared epitelial gastrointestinal y la destrucción del grosor de la mucosa intestinal (Marlicz y Loniewski, 2015), lo que desencadena una respuesta inmune inflamatoria. Estos procesos están asociados con daño celular y translocación de lipopolisacáridos (LPS) hacia afuera del tracto gastrointestinal, lo que en consecuencia desencadena respuestas inmunes e inflamatorias que a menudo resultan en un aumento en la permeabilidad intestinal (revisado por Clark y Mach, 2016) y también respuestas mitocondriales. Por ejemplo, estudios realizados en modelos animales han demostrado que la inyección de LPS (3 mg/kg) en gatos machos adultos provocaron un desacoplamiento parcial de la OXPHOS mitocondrial y una reducción del 40% de la actividad COX (Crouser y col. 2002), y también una mayor producción de ROS como resultado de una reducción en la expresión de la proteína de desacoplamiento 2 (UPC2; Saint- Georges-Chaumet et al., 2015). Lee y Hüttemann ( 2014) postularon que los TLR que se unen a LPS estimulan la producción de TNFα e IL-6 , quien activa la tirosin quinasa lo que provoca una fosforilación de COX corriente abajo y perjudica la producción de ATP en las mitocondrias.
Por otro lado, está surgiendo evidencia de que la actividad de SIRT1 puede atenuar las citoquinas inflamatorias inducidas por LPS y mejorar las funciones mitocondriales (Caruso et al ., 2014; Lo Sasso et al., 2014; Wei et al., 2017). Cheng et al. (2015) revelaron que el ejercicio activó la actividad SIRT1, lo que redujo la actividad inflamatoria de NF-κB, TLR-4, IL-1β, IL-6, IL-8 y TNFα. Un modelo de sepsis murina en ratones C57BL/6 inyectados con LPS observó la estimulación de TLR4 y la activación de SIRT1, lo que indujo la desacetilación y desactivación de NF-κB p65 inhibiendo la expresión de genes proinflamatorios (Liu et al. , 2012). SIRT1 podría desempeñar un papel importante en el mantenimiento de la función de la barrera intestinal a través de diversos mecanismos, tales como el incremento en la proliferación de criptas y supresión de la apoptosis de las vellosidades (Wang et al. 2012), la estimulación de la expansión de las células madre intestinales en el intestino (Igarashi y Guarente, 2016), la regulación de la expresión de zonulina ocludina-r1 y claudina-1 en las uniones estrechas durante hipoxia (Ma Y. et al. , 2014). SIRT1 también puede reducir la inflamación inducida por estrés y puede mejorar la acumulación de ROS inducida por isquemia /reperfusión intestinal y la apoptosis a través de la activación de miR-34a-5p , que induce la supresión de la acumulación de ROS intestinal mediada por SIRT1 (Wang et al. , 2016). Sin embargo, algunos estudios han demostrado que la señalización inflamatoria inducida por LPS puede inhibir la actividad SIRT1 (Shen et al. , 2009; Fernandes et al., 2012; Storka et al., 2013) sugiriendo que la actividad o inactividad de SIRT1 puede ser específica del tejido y podría depender de la dosis de LPS.
Otros estudios han demostrado que las citoquinas inflamatorias tales como el TNFα e IL -6 también pueden inhibir la activación de AMPK, lo que afecta negativamente el metabolismo de la glucosa y reduce la FAO en mitocondrias (Steinberg et al., 2006; Lim et al., 2010; Viollet et al., 2010; Andreasen et al., 2011). IL-6, que es otra citoquina asociada con las lesiones musculares inducidas por el ejercicio (Richter y Ruderman, 2009; Lantier et al., 2014) y con la permeabilidad intestinal (Grootjans et al. 2010) también puede activar AMPK en músculo esquelético tras el ejercicio prolongado (Febbraio y Pedersen , 2005; Ruderman et al. 2006), lo que podría mejorar el consumo periférico de glucosa y la sensibilidad a la insulina en todo el cuerpo (Glund et al. , 2007). Además, en corredores de maratón, la síntesis de IL-6 en el músculo esquelético puede aumentar hasta 100 veces (Febbraio y Pedersen, 2002). Ruderman y col. (2006) evaluaron los efectos metabólicos que tenía la IL-6 sobre la activación de AMPK durante el ejercicio en ratones knock-out IL-6. Llegaron a la conclusión de que los ratones maduros de 9 meses presentaban signos de dislipidemia, intolerancia a la glucosa y obesidad posiblemente debido a la falta de activación de AMPK inducida por IL-6. Kelly et al. (2009) analizaron la activación de AMPK inducida por IL-6 células del músculo esquelético de ratas in vivo inyectadas con 25 ng/g de peso animal. Los autores observaron que la IL-6 activa AMPK a través del aumento en la concentración de monofosfato de adenosina cíclica (cAMP) y de la relación AMP: ATP lo que favorece la síntesis de energía en los músculos. Otro estudio realizado por Lantier et al. (2014) demostró que los ratones knockout AMPKα1α2 tuvieron un rendimiento físico y una resistencia a la fatiga significativamente menores , así como también una mayor expresión de IL-6 posiblemente debido a lesiones en el músculo esquelético. Este estudio proporciona una clara evidencia de que la inactividad de AMPK redujo la fosforilación oxidativa (OXPHOS) mitocondrial lo que produjo una menor producción de energía en respuesta al ejercicio.
Subproductos de la microbiota comensal también pueden regular las respuestas inflamatorias mitocondriales a través de diferentes mecanismos durante el ejercicio de resistencia, siendo probablemente los principales la modulación de la producción de ROS en la barrera y la translocación de LPS. Por ejemplo, el indol 3-propionato producido por bacterias tiene efectos beneficiosos para el huésped porque mantiene la función de la barrera intestinal regulando por incremento a las proteínas de unión e inhibiendo la expresión de TNFα en enterocitos, lo que evita la translocación de LPS y de patógenos reduciendo así la respuesta inmunitaria inflamatoria (Boulangé y col. 2016). Los SCFA también regulan a la baja a los mediadores pro inflamatorios inducidos por LPS a través de la desacetilación de histonas en macrófagos en la lámina propia, y a los mediadores proinflamatorios tales como NO, IL-6 e IL-12 (Chang y col. 2014). Además, los SCFA inhiben la activación de NF-κB en macrófagos de lámina propia lo que provocó una disminución en la inflamación asociada con la colitis ulcerosa (Lührs et al. 2002) y también pueden regular el AMPK (Canfora et al. 2015) activando la vía de la UCP2-AMPK-acetil-CoA carboxilasa (ACC) (den Besten y col. 2015) y la proteína GPR43, quienes desempeñan un papel protector en la activación y en la señalización inflamatoria. Por ejemplo, Peng et al. (2009) determinaron que la línea celular del epitelio colónico humano Caco-2 tratada con 2 mmol/L de N-butirato presentó una regulación por incremento de la actividad de AMPK y una mayor resistencia transepitelial, un marcador de un correcto funcionamiento de la barrera intestinal. Sin embargo, no se ha establecido con detalle la activación de AMPK y su papel en la regulación de las proteínas de las uniones estrechas, aunque es probable que actúe a través de la fosforilación (Elamin y col. 2013). Vieira y col. (2015) observaron que el sensor del receptor metabólico GPR43 detectaba acetato en neutrófilos in vitro, se incrementaba la producción de IL-18, lo que favorecería la integridad del epitelio intestinal en modelos celulares para el estudio de colitis (Zaki y col. 2010; Macia et al., 2015). Del mismo modo, Macia et al. (2015) observaron que las dietas ricas en fibras aumentaban el acetato en el colon de ratones que presentaban colitis inducida por DSS y en consecuencia aumentaba la expresión de GPR43 y la secreción de IL-18. Por otro lado, una dieta que aumentaba los niveles de β-hidroxibutirato, un metabolito derivado de las cetonas inhibía la actividad de NLRP3 durante el ejercicio de alta intensidad (Shao y col. 2015; Youm y col. 2015).
Sin embargo, un entorno inflamatorio en el intestino podría constituir un ambiente favorable para la expansión de enterobacterias patógenas (Zeng y col. 2017). Las enterobacterias entre las que se incluyen E. coli, Klebsiella spp. y Proteus spp. se encuentran entre los patobiontes con mayor sobrecrecimiento más comúnmente observados en las afecciones inflamatorias (Zeng y col. 2017). La inflamación en el intestino puede provocar un aumento de oxígeno en la luz intestinal debido a que el flujo sanguíneo y la hemoglobina aumentan e inducen el crecimiento de patógenos en el intestino (Zeng y col. 2017). Otras bacterias, como la Clamidia pneumoníae (Shimada y col. 2012) y Salmonella typhimurium pueden inducir NLRP3 a través de la caspasa-11 de los macrófagos (de Zoete y Flavell, 2013) y pueden producir disfunción mitocondrial.
Cómo Regulan las Mitocondrias la Microbiota Intesinal
Lobet y col. (2015) describen que las mitocondrias participan en la detección de microorganismos infecciosos y del daño celular para activar respuestas inmunitarias innatas. Además, se demostró que las mitocondrias tienen un papel destacado en el modulación de las funciones intestinales (Igarashi y Guarente , 2016; Wang et al., 2016), tales como la protección de la barrera intestinal (Peng et al., 2009) y la respuesta inmune de la mucosa, todas fundamentales para el mantenimiento de la capa mucosa (Ma Y. y col. 2014) y de la microbiota intestinal (lShimada et al., 2012; Caron et al., 2014). Por lo tanto, la desregulación de las funciones mitocondriales puede afectar la microbiota intestinal a través de la alteración del entorno intestinal normal lo que permite que los antígenos bacterianos penetren en el epitelio y desencadenen la respuesta inmunitaria. Tal como se explicó anteriormente otra posible perturbación en el hábitat de la microbiota inducida por mitocondrias se produce a través de las modificaciones de las respuestas del sistema inmunitario.
Se ha demostrado que las variantes genéticas del genoma mitocondrial también pueden regular la microbiota intestinal. Hasta hace poco, la genética mitocondrial a menudo se consideraba fuera de los estudios principales de genética. La naturaleza poliploide del genoma mitocondrial (de incluso varios miles de copias por célula) da lugar a una característica importante de la genética mitocondrial, homoplasmia, y heteroplasmia. Algunas mutaciones afectan a todas las copias de genoma mitocondrial (mutación homoplasmática), mientras que otras solo están presentes en algunas copias del genoma mitocondrial (mutación heteroplasmática) (Taylor y Turnbull, 2005).
Los polimorfismos en los genes ND5 y CYTB o en la regíon D-Loop del genoma mitocondrial se han asociado con composiciones específicas de microbiota intestinal. Por ejemplo, Ma J. et al. ( 2014) demostraron que A13434G, un sinónimo SNP ubicado en el gen ND5 y el SNP sinónimo T15784C ubicado en CYTB se asociaron fuertemente con abundancia de especies de género Eubacterium (perteneciente al cluster IV de Clostridium) y Roseburia (cluster XIVa de Clostridium), que son anaerobios altamente sensibles al oxígeno y productores de butirato. De manera similar, el polimorfismo no sinónimo T14798C, que codifica los genes del citocromo b, se asoció fuertemente con abundancia diferencial de Dialister en el fórnix vaginal posterior (Evaldson et al. 1980). Estos hallazgos sugieren que las variantes del genoma mitocondrial del hospedador podrían definir inherentemente la composición y la función del microbioma intestinal, quien a su vez estructurará su comunidad.
Además, mutaciones del genoma nuclear que causan desequilibrios en las funciones mitocondriales (p. ej., mutación del gen TYMP que se observa en los pacientes con neuro gastro encefalomiopatía mitocondrial intestinal) o con trastornos en la oxidación de ácidos grasos de cadena larga en la mitocondria (p. ej., variantes patogénicas bialélicas de deficiencia de carnitina palmitoiltransferasa 1A) son más susceptibles a las infecciones bacterianas que la población general (Garone y col. 2011; Gessner et al., 2013). Adicionalmente, el haplogrupo europeo de ADN mitocondrial HV se ha asociado con una disminución de las probabilidades de sepsis grave, lo que sugiere que los haplotipos mitocondriales podrían determinar las tasas de supervivencia durante shock séptico severo debido a diferencias en su función (Baudouin et al., 2005; Lorente et al., 2012; Jiménez-Sousa y col. 2015), a una mayor capacidad de OXPHOS y mayor producción de ROS y RONS (Jiménez-Sousa y col. 2015). Más específicamente, Maruszak y col. (2014) informaron que los atletas olímpicos eran principalmente del haplogrupo HV, que se ha asociado con un VO2máx más alto en respuesta al ejercicio y una mayor capacidad de OXPHOS y, por lo tanto, una mayor producción aeróbica de ATP, mientras que un estudio realizado con varones caucásicos españoles sanos (n = 81) también demostró que el haplogrupo HV se asoció con una mayor producción de ROS y daño oxidativo mitocondrial en comparación a lo observado en al haplogrupo JT (Martinez y col. 2010).
Curiosamente, las mitocondrias se replican durante el ejercicio de resistencia, por lo que el ADN mitocondrial podría acumular mutaciones que eventualmente podrían comprometer la eficiencia de la OXPHOS y otras funciones esenciales, incluida la homeostasis intestinal (Green y col. 2011). Aunque, Safdar et al. (2011) demostraron que el ejercicio de resistencia redujo la frecuencia de mutaciones puntuales en ratones PolG, lo que produce un aumento concomitante en ensamblaje del complejo COX mitocondrial, la heteroplasmia mitocondrial se podría producir fácilmente en atletas de resistencia debido al hecho de que cada célula contiene entre 1000 y 100000 copias de ADN mitocondrial ( Khan et al., 2015), que a su vez pueden ser transmitidas a las generaciones subsiguientes (Stewart y Chinnery, 2015). Las divisiones celulares posteriores pueden dar lugar a una mayor o menor frecuencia de una determinada mutación, así como también mutaciones de novo durante la vida de un individuo (Stewart y Chinnery, 2015). Es necesario que se realicen estudios completos de genoma que logren caracterizar los genes mitocondriales específicos y las vías en el genoma humano nuclear y mitocondrial que puedan poner en evidencia la composición del microbioma de los atletas de resistencia.
Se necesitan más estudios para establecer como la heteroplasmia mitocondrial y las variantes genéticas nucleares afectan el funcionamiento de las mitocondrias y, a su vez, la composición y función de la microbiota. Se necesita un estudio holístico que busque comprender la dinámica compleja de la interacción entre el medio ambiente, la estocasticidad y los tres genomas (microbioma nuclear, mitocondrial y intestinal).
Conclusión
Muchas líneas de evidencia sugieren que las mitocondrias desempeñan un papel central en la producción de energía, producción de ROS y RONS y en la regulación de los inflamasomas durante el ejercicio de resistencia. El ejercicio de resistencia induce biogénesis mitocondrial sistémica, previene la degradación y las mutaciones del ADN mitocondrial, y aumenta la oxidación y la capacidad antioxidante mitocondrial. Sin embargo, el sobreentrenamiento y el estrés crónico promueven inflamación en el tracto gastrointestinal de atletas que da como resultado una gran cantidad de factores estresantes que favorecen la translocación de lipopolisacáridos y la proliferación de patobiontes. Estamos recién comenzando a comprender la comunicación bidireccional entre la microbiota intestinal y las mitocondrias, pero varios estudios han demostrado que las moléculas de la microbiota intestinal comensal, tales como el N-butirato, son esenciales para controlar el estrés oxidativo mitocondrial y la respuesta inflamatoria, el crecimiento y la adherencia de patógenos y también para mejorar el metabolismo y el gasto energético durante ejercicio. Además, los ácidos grasos de cadena corta pueden inducir mediadores importantes en la biogénesis mitocondrial a través de coactivadores transcripcionales tales como PCG-1α, del sensor de energía sensible a redox SIRT1 y la enzima AMPK, quienes suprimen la respuesta inflamatoria general y permiten la expresión de los efectos beneficiosos del ejercicio.
La disminución de la inflamación y el estrés oxidativo durante el ejercicio de resistencia sería un enfoque ideal para restringir el crecimiento desmedido de patobiontes en el intestino, así como sus efectos perjudiciales en las funciones mitocondriales. Aunque sigue siendo un desafío atenuar la respuesta inflamatoria a través de tratamientos dietéticos, el manejo de los cambios nutricionales y del estrés oxidativo podría ser un buen abordaje para mantener una microbiota intestinal saludable y por lo tanto mantener las funciones mitocondriales y la homeostasis del huésped. Por otro lado, la producción mitocondrial de ROS tiene un papel crucial en la regulación de las funciones intestinales, tales como la integridad de la barrera intestinal y las respuestas inmunitarias de la mucosa, que son importantes para regular la composición de la microbiota intestinal. A pesar del tamaño diminuto del genoma mitocondrial, las mutaciones del ADN mitocondrial se heredan y pueden afectar no solo funciones de los tejidos si no que también funciones de la microbiota. Las mutaciones en genes mitocondriales (por ej., ND5 y CYTB) o en la región D-loop y el estrés oxidativo también modulan la composición y las funciones de la microbiota intestinal. Por lo tanto, es necesario conocer las numerosas vías moleculares que conducen a la acumulación de mutaciones en el ADN mitocondrial
Contribuciones de los Autores
AC escribió el texto principal y tanto AC como NM diseñaron las figuras. NM aportó comentarios críticos sobre el contenido y el diseño y participó en la revisión del manuscrito. Ambos autores corrigieron y aprobaron la versión final del manuscrito.
Versión Original
Clark A. and Mach N. The Crosstalk between the Gut Microbiota and Mitochondria during Exercise.(2017). Frontiers in Physiology. Volume 8. Article 319.
REFERENCIAS
Ahmad,M.S.,Krishnan,S.,Ramakrishna,B.S.,Mathan,M.,Pulimood,A.B.,andMurthy,S.N.(2000) Butyrate and glucose metabolism by colonocytes in experimental colitis in mice. Gut 46,493–499.
Alex,S.,Lichtenstein,L.,Dijk,W.,Mensink,R.P.,Tan,N.S.,and Kersten,S.(2014).ANGPTL4is produced by entero-endocrine cells in the human intestinal tract. Histochem.CellBiol. 141,383–391. doi:10.1007/s00418-013-1157-y
Andersson,S.G.E.,Zomorodipour,A.,Andersson,J.O.,Sicheritz-Pontén,T., Alsmark,U.C.M.,Podowski,R.M.,et al.(1998).The genome sequence of Rickettsia prowazekii and the origin of mitochondria. Nature 396,133–140.
Andreasen,A.S.,Kelly,M.,Berg,R.M.G.,Møller,K.,andPedersen,B.K.(2011).Type2 Diabetes Is Associated with Altered NF-κB DNA Binding Activity, JNK Phosphorylation, and AMPK Phosphorylation in Skeletal Muscleafter LPS.PLoSONE 6:e23999.doi:10.1371/journal.pone.0023999
Bäckhed,F.,Manchester,J.K.,Semenkovich,C.F.,andGordon,J.I.(2007). Mechanisms underlying the resistance to diet-induced obesity in germ-free mice. Proc.Natl.Acad.Sci.U.S.A. 104,979–984.doi:10.1073/pnas.0605374104
Bär,F.,Bochmann,W.,Widok,A.,Medem,K.,vonPagel,R.,Hirose, M.,et al. (2013). Mitochondrial gene polymorphisms that protect mice from colitis. Gastroenterology 145,1055 1063.e3.doi:10.1053/j.gastro.2013.07.015
Bartlett,K.,and Eaton,S.(2004).Mitochondrial β-oxidation. Euro.J.Biochem. 271, 462 469.doi:10.1046/j.1432-1033.2003.03947.x
Baudouin,S.V.,Saunders,D.,Tiangyou,W.,Elson,J.L.,Poynter,J.,Pyle,A.,
etal.(2005).Mitochondrial DNA and survival after sepsis:a prospective study. Lancet 366,2118–2121.doi:10.1016/S0140-6736(05)67890-7
Beaumont,M.,Andriamihaja,M.,Lan,A.,Khodorova,N.,Audebert,M.,Blouin, J.-M.,etal.(2016).Detrimental effects for colonocytes of an increased exposure to luminal hydrogen sulfide:thea daptive response. FreeRadic.Biol.Med. 93,155–164.doi:10.1016/j.freeradbiomed.2016.01.028
Befroy, D.E.,Petersen,K.F.,Dufour,S.,Mason,G.F.,Rothman,D.L.,and Shulman,G.I.(2008).Increased substrate oxidation and mitocondrial Uncoupling in skeletal muscle of endurance-trained individuals. Proc.Natl. Acad.Sci.U.S.A. 105,16701–16706.doi:10.1073/pnas.0808889105
Blachier,F.,Mariotti,F.,Huneau,J.F.,andTomé,D.(2007).Effects of aminoacid-derived luminal metabolites on the colonic epithelium and physiopathological consequences. AminoAcids 33,547–562.doi:10.1007/s00726-006-0477-9
Blaser,H.,Dostert,C.,Mak,T.W.,andBrenner,D.(2016).TNFandROSCrosstalk in Inflammation. Trends Cell Biol. 26,249–261.doi:10.1016/j.tcb.2015.12.002
Bloomer,R.J.,Goldfarb,A.H.,Wideman,L.,McKenzie,M.J.,and Consitt,L.A.(2005).Effects of acute aerobic and anaerobic exercise on Blood markers of oxidative stress. J.StrengthCond.Res. 19,276–285. doi:10.1519/00124278-200505000-00007
Bo,H.,Zhang,Y.,andJi,L.L.(2010).Redefining the role of mitochondria in exercise: a dynamic remodeling. Ann.N.Y.Acad.Sci. 1201,121–128. doi:10.1111/j.1749-6632.2010.05618.x
Boulangé,C.L.,Neves,A.L.,Chilloux,J.,Nicholson,J.K.,and Dumas,M.-E. (2016).Impact of the gut microbiota on inflammation, obesity, and metabolic disease. GenomeMed 8:42.doi:10.1186/s13073-016-0303-2
Busquets-Cortés,C.,Capó,X.,Martorell,M.,Tur,J.A.,Sureda,A., and Pons,A.(2016).Training enhances immune cells mitocondrial biosynthesis, fission, fusion, and their antioxidant capabilities synergistically with dietary
Docosahexaenoic supplementation. Oxid.Med.CellLongev 2016:8950384. doi:10.1155/2016/8950384
Canfora,E.E.,Jocken,J.W.,andBlaak,E.E.(2015).Short-chain fatty acids in control of body weight and insulin sensitivity. Nat.Rev.Endocrinol. 11, 577–591.doi:10.1038/nrendo.2015.128
Caron,A.Z.,He,X.,Mottawea,W.,Seifert,E.L.,Jardine,K.,Dewar-Darch,D., et al.(2014).The SIRT1 deacetylase protects micea gainst the symptoms of Metabolic syndrome. FASEBJ. 28,1306–1316.doi:10.1096/fj.13-243568
Caruso,R.,Marafini,I.,Franzè,E.,Stolfi,C.,Zorzi,F.,Monteleone,I.,etal.(2014). Defective expression of SIRT1contributes to sustain inflammatory pathways in The gut. MucosalImmunol. 7,1467–1479.doi:10.1038/mi.2014.35
Chang,P.V.,Hao,L.,Offermanns,S.,andMedzhitov,R.(2014). The Microbial metabolite butyrate regulates intestinal macrophage function via Histone deacetylase inhibition. Proc.Natl.Acad.Sci.U.S.A. 111,2247–2252.
doi:10.1073/pnas.1322269111
Cheng,Y.-Y.,Kao,C.-L.,Ma,H.-I.,Hung,C.-H.,Wang,C.-T.,Liu,D.-H.,et al.(2015).SIRT1-related inhibition of pro-inflammatory responses and oxidative Stress are involved in the mechanism of nonspecific lowback pain relief after exercise through modulation of Toll-like receptor 4. J.Biochem. 158,299–308. doi:10.1093/jb/mvv041
Cheng,Z.,andRistow,M.(2013).Mitochondria and metabolic homeostasis. Antioxid. Redox Signal. 19,240–242.doi:10.1089/ars.2013.5255
Chiang,S.M.,andSchellhorn,H.E.(2012).Regulators of oxidative stress response genes in Escherichia coli and their functional conservation in bacteria. Arch. Biochem. Biophys. 525,161–169.doi:10.1016/j.abb.2012.02.007
Circu,M.L.,andAw,T.Y.(2012).Intestinal redox biology and oxidative stress. Semin. Cell Dev.Biol. 23,729–737.doi:10.1016/j.semcdb.2012.03.014
Clark,A.,andMach,N.(2016).Exercise-induced stress behavior, gut-microbiota-
Brain axis and diet:a systematic review for athletes. J.Int.Soc.SportsNutr. 13:43.doi:10.1186/s12970-016-0155-6
Clarke,S.F.,Murphy,E.F.,O’Sullivan,O.,Lucey,A.J.,Humphreys,M.,Hogan,A., Et al.(2014).Exercise and associated dietary extremes impact on gut microbial diversity. Gut 63,1913–1920.doi:10.1136/gutjnl-2013-306541
Crane,J.D., Abadi,A.,Hettinga,B.P.,Ogborn,D.I.,MacNeil,L.G.,
Steinberg,G.R.,et al.(2013).Elevated mitocondrial oxidative stress impairs metabolic adaptations to exercise in skeletal muscle. PLoSONE 8:81879. doi:10.1371/journal.pone.0081879
Crouser,E.D.,Julian,M.W.,Blaho,D.V.,andPfeiffer,D.R.(2002).Endotoxin-
Induced mitocondrial damage correlates with impaired respiratory activity. Crit.CareMed. 30,276–284.doi:10.1097/00003246-200202000- 00002
Cunningham,K.E.,Vincent,G.,Sodhi,C.P.,Novak,E.A.,Ranganathan,S.,Egan, C.E.,et al.(2016).Peroxisome Proliferator-activated Receptor-γ Coactivator 1-α (PGC1α). Protects against Experimental Murine Colitis. J.Biol.Chem. 291, 10184–10200.doi:10.1074/jbc.M115.688812
Den Besten,G.,Gerding,A.,vanDijk,T.H.,Ciapaite,J.,Bleeker,A.,van Eunen,K.,et al.(2015).Protection against the Metabolic Syndrome b yGuar Gum-Derived Short-Chain Fatty Acids Depends on Peroxisome Proliferator- Activated Receptor γ and Glucagon-LikePeptide-1. PLoSONE 10:136364. doi:10.1371/journal.pone.0136364
denBesten,G.,van Eunen,K.,Groen,A.K.,Venema,K.,Reijngoud,D.-J.,and Bakker,B.M.(2013).The role of short-chain fatty acids in the interplay between diet gut microbiota and host energy metabolism. J.Lipid.Res. 54,2325–2340. doi:10.1194/jlr.R036012
deZoete,M.R.,andFlavell,R.A.(2013).Interactions between Nod-Like Receptors and Intestinal Bacteria. Front.Immunol. 4:462.doi:10.3389/fimmu.2013.00462
Dobashi,Y.,Miyakawa,Y.,Yamamoto,I.,andAmao,H.(2011).Effects of intestinal microflora on superoxide dismutase activity in the mouse cecum. Exp.Animals 60,133–139.doi:10.1538/expanim.60.133
Dobashi,Y.,Yoshimura,H.,Atarashi,E.,Takahashi,K.,Tohei, A., and Amao,H. (2013).Up regulation of superoxide dismutase activity in the intestinal tract Mucosa of Germ-FreeMice. J.Veter. Med.Sci. 75,49–54.
doi:10.1292/jvms.12-0248
Donohoe,D.R.,Garge,N.,Zhang,X.,Sun,W.,O’Connell,T.M.,Bunger, M.K.,etal.(2011).The Microbiome and Butyrate Regulate Energy Metabolism and Autophagy in the Mammalian Colon. CellMetab. 13,517–526.
doi:10.1016/j.cmet.2011.02.018
Duncan,S.H.,Louis,P.,andFlint,H.J.(2004).Lactate-utilizing Bacteria ,isolated from human feces, that produce butyrate as a Major fermentation product. Appl.Environ.Microbiol. 70,5810–5817.
doi:10.1128/AEM.70.10.5810-5817.2004
Elamin,E.E.,Masclee,A.A.,Dekker,J.,Pieters,H.-J.,andJonkers,D.M.(2013). Short-chain fatty acids activateAMP-activated protein kinase and ameliorate ethanol-induced intestinal barrier dysfunction in Caco-2 Cell Monolayers. J. Nutr. 143,1872–1881.doi:10.3945/jn.113.179549
Evaldson,G.,Carlström,G.,Lagrelius,A.,Malmborg,A.-S.,andNord, C.E.(1980). Microbiological findings in pregnant women with premature ruptura of the membranes. Med. Microbiol. Immunol. 168,283–297.doi:10.1007/BF02121812
Febbraio,M.A., and Pedersen,B.K.(2002).Muscle derived interleukin-6: mechanisms for activation and posible biological roles. FASEBJ. 16, 1335–1347.doi:10.1096/fj.01-0876rev
Febbraio,M.A.,and Pedersen,B.K.(2005).Contraction-induced myokine Production and release: isskeletal muscle an endocrine organ? Exerc.SportSci. Rev. 33,114–119.doi:10.1097/00003677-200507000-00003
Fernandes,C.A.,Fievez,L.,Neyrinck,A.M.,Delzenne,N.M., Bureau, F., and Vanbever,R.(2012).Sirtuin inhibition attenuates the production of inflammatory cytokines in lipopolysaccharide stimulated macrophages.
Biochem.Biophys.Res.Commun. 420,857–861.doi:10.1016/j.bbrc.2012.03.088
Fisher-Wellman,K.,and Bloomer,R.J.(2009).Acute exercise and oxidative stress: A 30 year history. Dyn.Med. 8:1.doi:10.1186/1476-5918-8-1
Flint,H.J., Bayer,E.A. ,Rincon,M.T., Lamed,R.,and White,B. A.(2008). Polysaccharide utilization by gut bacteria: potential for new insights from Genomic analysis. Nat.Rev.Microbiol. 6,121–131.doi:10.1038/nrmicro1817
Gao,Z.,Yin,J.,Zhang,J.,Ward,R.E.,Martin,R.J.,Lefevre,M.,etal.(2009). Butyrate improves insulin sensitivity and increases energy expenditure in mice. Diabetes 58,1509–1517.doi:10.2337/db08-1637
Garatachea,N., Pareja-Galeano,H., Sanchis-Gomar,F., Santos-Lozano,A., Fiuza-Luces, C. , Morán,M., et al.(2015). Exercise attenuates the major hallmarks of aging. Rejuven. Res. 18,57–89.doi:10.1089/rej.2014.1623
Garone,C., Tadesse,S.,and Hirano,M. (2011).Clinical and genetic Spectrum of mitocondrial neurogastrointestinal encephalomyopathy. Brain 134,3326–3332.doi:10.1093/brain/awr245
Gérard,P.(2013).Metabolism of Cholesterol and Bile Acids by the Gut Microbiota. Pathogens 3,14–24.doi:10.3390/pathogens3010014
Gessner,B.D.,Gillingham,M.B.,Wood,T.,andKoeller,D.M.(2013). Association of a GeneticVariant of Carnitine Palmitoyl transferase1A With Infections in Alaska Native Children. J.Pediatr. 163,1716–1721.
doi:10.1016/j.jpeds.2013.07.010
Ghosh,S.,Dai,C.,Brown,K.,Rajendiran,E.,Makarenko,S.,Baker,J., Et al.(2011). Colonic microbiota alters host susceptibility to infectious Colitis by modulating Cammation, redox status and ion transporter Gene expression. Am.J. Physiol. Gastrointest.Liver Physiol. 301,G 39–G49. doi:10.1152/ajpgi.00509.2010
Glund,S.,Deshmukh,A.,Long,Y.C.,Moller,T., Koistinen,H.A., Caidahl,K.,etal. (2007).Interleukin-6 directly increases glucose metabolism in resting human Skeletal muscle. Diabetes 56,1630–1637.doi:10.2337/db06-1733
Gomes, E.C. Silva,A.N.,and de Oliveira,M.R.(2012).Oxidants,antioxidants, And the beneficial roles of exercise induced production of reactive species. Oxid. Med. Cell Longev. 2012:756132.doi:10.1155/2012/756132
Gray, M.W., Burger,G.,and Lang,B.F.(1999).Mitochondrial evolution. Science 283,1476–1481.
Gray, M.W., Burger,G.,andLang,B.F.(2001).Theorigin and Early evolution of mitochondria. Genome Biology 2:reviews1018. doi:10.1186/gb-2001-2-6-reviews1018
Green, D.R., Galluzzi,L.,and Kroemer,G.(2011).Mitochondria and the
Autophagy inflammation–cell death axis in organismal aging. Science 333, 1109–1112.doi:10.1126/science.1201940
Grootjans,J.,Lenaerts,K.,Derikx,J.P.M.,Matthijsen,R.A.,Bruïne,A.P., De Bijnen,A.A.,et al.(2010).Human Intestinal Ischemia-Reperfusion– Induced Inflammation Characterized. Am.J.Pathol. 176,2283–2291.
doi:10.2353/ajpath.2010.091069
Hamer,H.M., Jonkers,D.M.A.E., Bast,A.,Vanhoutvin, S.A.L. W., Fischer,M.A.J.G.,Kodde,A.,et al.(2009).Butyrate modulates oxidative Stress in the colonic mucosa of healthy humans. Clin.Nutr. 28,88–93.
doi:10.1016/j.clnu.2008.11.002
Holland,A.M.,Hyatt,H.W.,Smuder,A.J.,Sollanek,K.J.,Morton,A.B.,Roberts, M.D.,et al.(2015).Influence of endurance exercise training on antioxidant
enzymes, tight junction proteins, and inflammatory markers in the rat ileum. BMC Res Notes 8:6.doi:10.1186/s13104-015-1500-6
Hood,D.A.(2001).Invited Review:Contractile activity-induced mitocondrial Biogénesis in skeletal muscle. J. Appl. Physiol. 90,1137–1157.
Hood,D.A.,Uguccioni,G.,Vainshtein,A.,and D’souza,D.(2011).Mechanisms of exercise-induced mitocondrial biogénesis in skeletal muscle: implications for health and disease. Compr. Physiol. 1,1119–1134.doi:10.1002/cphy.c100074
Hooper,L.V.,and Gordon,J.I.(2001).Commensal host-bacterial relationships in The gut. Science 292,1115–1118.doi:10.1126/science.1058709
Hsu,Y.J.,Chiu,C.C.,Li,Y.P.,Huang,W.C.,Huang,Y.T.,Huang,C.C., et al.(2015).Effect of intestinal microbiota on exercise performance in mice.J. Strength Cond. Res. 29,552–558.doi:10.1519/JSC.0000000000000644
Huang,C.-C.,Wang,T.,Tung,Y.-T.,andLin,W.-T.(2016).Effect of exercise Training on skeletal muscle SIRT1 and PGC-1αexpression levels in rats of different age. Int.J.Med.Sci. 13,260–270.doi:10.7150/ijms.14586
Hylemon,P.B.,Zhou,H.,Pandak,W.M.,Ren,S.,Gil,G.,andDent,P. (2009).Bile acids as regulatory molecules. J.Lipid.Res. 50,1509–1520. doi:10.1194/jlr.R900007-JLR200
Igarashi,M.,and Guarente,L.(2016). mTORC1and SIRT1cooperate to fosterExpansión of gut adult stem cells during caloric restriction. Cell 166,436–450. doi:10.1016/j.cell.2016.05.044
Ito,Y.,Yonekura,R.,Maruta,K.,Koike,T.,Nakagami,Y.,Shibata,K.,etal.(2003). Tryptophan metabolism was accelerated by exercise in rat. Adv.Exp.Med.Biol.527,531–535.doi:10.1007/978-1-4615-0135-061
Jacouton,E.,Mach,N.,Cadiou,J.,Lapaque,N.,Clément,K.,Doré,J.,etal. (2015). Lactobacillus rhamnosus CNCMI-4317Modulates Fiaf/Angptl4 in Intestinal epitelial cells and circulating level in mice. PLoSONE 10:e0138880. doi:10.1371/journal.pone.0138880
Jiménez-Sousa,M.A.,Tamayo,E.,Guzmán-Fulgencio,M.,Heredia, M., Fernández-Rodríguez,A.,Gómez,E.,et al.(2015). Mitochondrial DNA Haplo groups are associated with severe sepsis and mortality in patients who
Underwent major surgery. J. Infect. 70,20–29.doi:10.1016/j.jinf.2014.07.005
Joyce,S.A.,and Gahn,C.G.M.(2016).Bile Acid Modifications at the Microbe-Host Interface:potential for Nutraceutical and Pharmaceutical Interventions in Host Health. Annu. Rev. Food Sci.Technol. 7,313–333.
doi:10.1146/annurev-food-041715-033159
Joyner,M.J.,and Coyle,E.F.(2008).Endurance exercise performance:the physiology of champions. J.Physiol. 586,35–44. doi:10.1113/jphysiol.2007.143834
Kazgan,N.,Metukuri,M.R.,Purushotham,A.,Lu,J.,Rao,A.,Lee,S.,et al.(2014). Intestine-specific deletion of Sirt1 in mice impairs DCoH2–HNF1α–FXR Signaling and alters systemic bile acid homeostasis. Gastroenterology 146, 1006–1016.doi:10.1053/j.gastro.2013.12.029
Kelly,M.,Gauthier,M.-S.,Saha,A.K.,andRuderman,N.B.(2009).Activation Of AMP-activated protein kinase by interleukin-6 in rat skeletal muscle: Association with changes in cAMP,energy state, and endogenous fuel
mobilization. Diabetes 58,1953–1960.doi:10.2337/db08-1293
Khan,N.A.,Govindaraj,P.,Meena,A.K.,andThangaraj,K.(2015).Mitochondrial disorders: challenges in diagnosis and treatment. Indian J. Med. Res. 141,13–26. doi:10.4103/0971-5916.154489
Kim,J.-H.,Kim,H.K.,Ko,J.-H.,Bang,H.,andLee,D.-C.(2013).The Relationship between Leukocyte Mitochondrial DNACopy number and Telomere length in community-dwelling elderly women. PLoSONE 8:e67227. doi:10.1371/journal.pone.0067227
Kimura,I.,Inoue,D.,Hirano,K.,andTsujimoto,G.(2014).TheSCFA Receptor GPR43 and energy metabolism. Front.Endocrinol.(Lausanne). 5:85. doi:10.3389/fendo.2014.00085
Knuiman,P.,Hopman,M.T.E.,and Mensink,M.(2015).Glycogen availability And skeletal muscle adaptations with endurance and resistance exercise. Nutr. Metab.(Lond). 12:59.doi:10.1186/s12986-015-0055-9
Korecka,A.,Wouters,T.,de,Cultrone,A.,Lapaque,N.,Pettersson,S.,Doré,J.,
Et al.(2013).ANGPTL4expression induced by butyrate and rosiglitazone in Human intestinal epitelial cells utilized in dependent pathways. Am.J.Physiol. Gastrointest. Liver Physiol. 304,G1025–G1037.doi:10.1152/ajpgi.00293.2012
Kuipers,F.,Bloks,V.W.,andGroen,A.K.(2014).Beyond intestinal soap–bile acids in metabolic control. Nat.Rev.Endocrinol. 10,488–498. doi:10.1038/nrendo.2014.60
Kumavath,R.N.,Barh,D.,Azevedo,V.,andKumar,A.P.(2017). Potential pharmacological applications of enzymes associated with bacterial metabolism Of aromatic compounds. JMA 9,1–13.doi:10.5897/JMA2015.0354
Kurland,C.G.,and Andersson,S.G.E.(2000).Origin and evolution Of the mitocondrial proteome. Microbiol.Mol.Biol.Rev. 64,786–820. doi:10.1128/MMBR.64.4.786-820.2000
Kurnasov,O.,Goral,V.,Colabroy,K.,Gerdes,S.,Anantha,S.,Osterman, A.,et al.(2003).NAD Biosynthesis. Chem.Biol. 10,1195–1204. doi:10.1016/j.chembiol.2003.11.011
Lakhan,S.E.,andKirchgessner,A.(2010).Gut inflammation in chronic fatigue syndrome. Nutr.Metab.(Lond). 7:79.doi:10.1186/1743-7075-7-79
Lantier,L.,Fentz,J.,Mounier,R.,Leclerc,J.,Treebak,J.T.,Pehmøller, C.,et al.(2014).AMPK controls exercise endurance, mitocondrial Oxidative capacity,and skeletal muscle integrity. FASEBJ. 28,3211–3224. doi:10.1096/fj.14-250449
Le Chatelier,E.,Nielsen,T.,Qin,J.,Prifti,E.,Hildebrand,F., Falony,G.,et al. (2013).Richness of human gut microbiome correlates with metabolic markers. Nature 500,541–546.doi:10.1038/nature12506
Lee,H.-C.,and Wei,Y.-H.(2005).Mitochondrial biogénesis and mitocondrial DNA maintenance of mammalian cells under oxidative stress. Int.J.Biochem. Cell Biol. 37,822–834.doi:10.1016/j.biocel.2004.09.010
Lee,I.,and Hüttemann,M.(2014). Energy crisis: the role of oxidative Phosphorylation in acute inflammation and sepsis. Biochim.Biophys.Acta 1842, 1579–1586.doi:10.1016/j.bbadis.2014.05.031
Leschelle,X.,Goubern,M.,Andriamihaja,M., Blottière,H.M., Couplan, E., Gonzalez-Barroso,M.-M.,et al.(2005).Adaptative metabolic response Of human colonic epitelial cells to the adverse effects of the luminal
Compound sulfide. Biochim. Biophys. Acta Gen. Subjects 1725,201–212. doi:10.1016/j.bbagen.2005.06.002
Li,H.,Miao,W.,Ma,J.,Xv,Z.,Bo,H.,Li,J.,etal.(2015).Acute exercise-induced Mitocondrial stress triggers an inflammatory response in the Myocardium Via NLRP3 inflammasome activation with mitophagy. Oxid.Med.Cell.Longev. 2016:e1987149.doi:10.1155/2016/1987149
Li,J.,Jia,H.,Cai,X.,Zhong,H.,Feng,Q.,Sunagawa,S.,et al.(2014).An integrated catalog of reference genes in the human gut microbiome. Nat.Biotechnol. 32, 834–841.doi:10.1038/nbt.2942.
Lim,C.T.,Kola,B.,andKorbonits,M.(2010).AMPK as a mediator of hormonal signalling. J. Mol. Endocrinol. 44,87–97.doi:10.1677/JME-09-0063.
Lira,V.A.,Benton,C.R.,Yan,Z.,andBonen,A.(2010).PGC-1α regulation by exercise training and its influences on muscle function and Insulin sensitivity. Am. J. Physiol. Endocrinol. Metab. 299, E145–E161. doi:10.1152/ajpendo.00755.2009
Little,J.P.,Safdar,A.,Cermak,N.,Tarnopolsky,M.A.,and Gibala,M.J.(2010). Acute endurance exercise increases the nuclear abundance of PGC-1α in Trained human skeletal muscle. Am. J. Physiol. Regul. Integr. Compar.Physiol. 298,R912–R917.doi:10.1152/ajpregu.00409.2009
Liu,T.F.,Vachharajani,V.T.,Yoza,B.K.,and McCall,C.E.(2012). NAD+-dependent Sirtuin1 and 6 proteins coordinate as witch fromglucose To fatty acid oxidation during the acute inflammatory response. J. Biol. Chem.
287,25758–25769.doi:10.1074/jbc.M112.362343
Lobet, E.,Letesson,J.-J.,andArnould,T.(2015).Mitochondria:a target for bacteria. Biochem.Pharmacol. 94,173–185.doi:10.1016/j.bcp.2015.02.007
Lorente,L.,Iceta,R.,Martín,M.M.,López-Gallardo,E.,Solé-Violán,J.,Blanquer, J.,et al.(2012).Survival and mitocondrial function in septic patients according To mitocondrial DNA haplogroup. Crit. Care 16:R10.doi:10.1186/cc11150
LoSasso,G.,Ryu,D.,Mouchiroud,L.,Fernando,S.C.,Anderson,C.L.,Katsyuba,
E.,et al.(2014).Loss of Sirt1 function improves intestinal anti-bacterial defense and protects from colitis-induced colorectal cancer. PLoSONE 9:102495. doi:10.1371/journal.pone.0102495
Lozupone,C.A.,Stombaugh,J.I.,Gordon,J.I.,Jansson,J.K., and Knight,R. (2012). Diversity,stability and resilience of the human gut microbiota. Nature 489,220–230.doi:10.1038/nature11550
Lührs,H.,Gerke,T.,Müller,J.G.,Melcher,R.,Schauber,J.,Boxberge,F.,etal. (2002).Butyrate inhibits NF-κB activation in lamina propia macrophages of patients with ulcerative colitis. Scand. J. Gastroenterol. 37,458–466. doi:10.1080/003655202317316105
Lundby,C.,and Jacobs,R.A.(2016).Adaptations of skeletal muscle mitocondria to exercise training. Exp.Physiol. 101,17–22.doi:10.1113/EP085319
Ma,J.,Coarfa,C.,Qin,X.,Bonnen,P.E.,Milosavljevic,A.,Versalovic,J., Et al.(2014).mt DNA haplo group and single nucleotide polymorphisms structure human microbiome communities. BMCGenomics 15:257.
doi:10.1186/1471-2164-15-257
Ma,Y.,Xu,C.,Wang,W.,Sun,L.,Yang,S.,Lu,D.,et al.(2014). Role Of SIRT1 in the protection of intestinal epitelial barrier under hipoxia and its mechanism. ZhonghuaWeiChangWaiKeZaZhi 17,602–606. doi:10.1371/journal.pone.0138307
Mach,N.,and Fuster-Botella,D.(inpress).Endurance exercise and gut microbiota: A review. J.SportHealthSci. doi:10.1016/j.jshs.2016.05.001
Mach,N.,Plancade,S.,Pacholewska,A.,Lecardonnel,J.,Rivière,J.,Moroldo, M., .(2016).Integrated mRNA and miRNA expression profiling in blood reveals candidate biomarker sassociated with endurance exercise in the horse. Sci.Rep. 6:22932.doi:10.1038/srep22932
Mach,N.,Ramayo-Caldas,Y.,Clark,A.,Moroldo,M.,Robert,C.,Barrey,E.,et al.
(2017).Understanding the response to endurance exercise using a systems biology approach:combining blood metabolomics,transcriptomics and miRNomics in horses. BMC Genomics 18:187.doi:10.1186/s12864-017-3571-3
Macia,L.,Tan,J.,Vieira,A.T.,Leach,K.,Stanley,D.,Luong,S.,et al.(2015). Metabolite-sensing receptors GPR43 and GPR109. A facilitate dietary fibre- Induced gut homeostasis through regulation of the inflammasome. Nat.
Commun. 6:6734.doi:10.1002/art.39107
Magalhães,J., Falcão-Pires,I., Gonçalves,I.O., Lumini-Oliveira,J.,Marques- Aleixo,I., dos Passos, E. ,et al.(2013).Synergistic impact of endurance Training and intermittent hypobaric hipoxia on cardiac function and
Mitocondrial energetic and signaling. Int.J.Cardiol. 168,5363–5371. doi:10.1016/j.ijcard.2013.08.001
Marley,M.G.,Meganathan,R.,and Bentley,R.(1986).Menaquinone (vitamin K2) biosynthesisin Escherichia coli: synthesis of o-succinyl benzoate does not Require the decarboxylase activity of theketoglutarate dehydrogenase complex. Biochemistry 25,1304–1307.
Marlicz,W.,and Loniewski,I.(2015).The effect of exercise and diet on gut Microbial diversity. Gut 64,519–520.doi:10.1136/gutjnl-2014-307909
Martinez,C.A.R.,Ribeiro,M.L.,Gambero,A.,Miranda,D.D.,da Pereira,C. J.A.,and Nadal,S.R.(2010).The importance of oxygen free radicals in the Etiopathogenesis of diversión colitis in rats. Acta Cirurg.Brasil. 25,387–395. doi:10.1590/S0102-86502010000500002
Maruszak,A., Adamczyk,J.G., Siewierski,M.,Soza´ nski,H.,Gajewski,A., and Å˙ zekanowski,C.(2014).Mitochondrial DNA variation is associated with elite Athletic status in the Polish population. Scand. J. Med. Sci. Sports 24,311–318. doi:10.1111/sms.12012
Meissner,M.,Lombardo,E.,Havinga,R.,Tietge,U.J.F.,Kuipers,F.,and Groen,A.K.(2011).Voluntary Wheel running increases bile acid as well as Colesterol excretion and decreases aterosclerosis in hypercholesterolemic mice. Atherosclerosis 218,323–329.doi:10.1016/j.atherosclerosis.2011.06.040
Mollica,M.P.,Raso,G.M.,Cavaliere,G.,Trinchese,G.,DeFilippo, C., Aceto,S.,et al.(2017).Butyrate regulates liver mitocondrial function, efficiency and dynamic ininsulin resistan obese mice. Diabetes 66,1405–1418.
doi:10.2337/db16-0924
Mottawea,W.,Chiang,C.-K.,Mühlbauer,M.,Starr,A.E.,Butcher,J.,Abujamel, T.,et al.(2016).Altered intestinal microbiota–host mitocondria crosstalk in New onset Crohn’s disease. Nat.Commun. 7:13419.doi:10.1038/ncomms13419
Neis,E.P.J.G.,Dejong,C.H.C.,and Rensen,S.S.(2015).The roleof microbial aminoacid metabolism in host metabolism. Nutrients 7,2930–2946. doi:10.3390/nu7042930
Nicholson,J.K.,Holmes,E.,Kinross,J.,Burcelin,R.,Gibson,G.,Jia,W.,et al. (2012). Host-gut microbiota metabolic interactions. Science 336,1262–1267. doi:10.1126/science.1223813
Nie,Y.,Hu,J.,andYan,X.(2015).Cross-talk between bile acids and intestinal Microbiota in host metabolism and health. J.ZhejiangUniv.Sci.B 16,436–446. doi:10.1631/jzus.B1400327
O’Callaghan,N.J.,Toden,S.,Bird,A.R.,Topping,D.L.,Fenech, M.,and Conlon, M.A.(2012).Colonocyte telomere shortening is greater with dietary red meat tan White meat and is attenuated by resistant starch. Clin.Nutr. 31,60–64. doi:10.1016/j.clnu.2011.09.003
Palazzetti,S.,Richard,M.-J.,Favier,A.,andMargaritis,I. (2003).Overloaded Training increases exercise-induced oxidative stress and damage. Can.J.Appl. Physiol. 28,588–604.
Papa,S.,Martino,P.L.,Capitanio,G.,Gaballo,A.,De Rasmo,D., Signorile,A.,et al.(2012).The oxidative phosphorylation system In mammalian mitochondria. Adv.Exp.Med.Biol. 942,3–37. doi:10.1007/978-94-007-2869-1_1
Peng,L.,Li,Z.-R.,Green,R.S.,Holzman,I.R.,andLin,J.(2009).Butyrate Enhances the intestinal barrier by facilitating tight junction assembly via Activation of AMP-Activated Protein Kinase in Caco-2 Cel lMonolayers. J.Nutr. 139,1619–1625.doi:10.3945/jn.109.104638
Pilegaard,H.,Saltin,B.,and Neufer,P.D.(2003).Exercise induces transient transcriptional activation of thePGC-1αgeneinhumanskeletalmuscle. J. Physiol. 546(Pt3),851–858.doi:10.1113/jphysiol.2002.034850
Pyne,D.B.,West,N.P.,Cox,A.J.,and Cripps,A.W.(2015).Probiotics Supplementation for athletes-clinical and physiological effects. Eur.J.Sport Sci. 15,63–72.doi:10.1080/17461391.2014.971879
Radak,Z.,Chung,H.Y.,Koltai,E.,Taylor,A.W.,and Goto,S.(2008). Exercise oxidative stress and hormesis. Ageing. Res.Rev. 7,34–42. doi:10.1016/j.arr.2007.04.004
Radak,Z.,Zhao,Z.,Koltai,E.,Ohno,H.,and Atalay,M.(2013). Oxygen consumption and usage during physical exercise:the balance between oxidative Stress and ROS-dependent adaptive signaling. Antioxid Redox Signal 18, 1208–1246.doi:10.1089/ars.2011.4498
Rajili´ c-Stojanovi´ c,M.,anddeVos,W.M.(2014).The first1000 cultured species of the human gastrointestinal microbiota. FEMS Microbiol. Rev. 38,996–1047. doi:10.1111/1574-6976.12075
Rapoport,B.I.(2010).Metabolic factors limiting performance in maratón runners. PLoS Comput.Biol. 6:e1000960.doi:10.1371/journal.pcbi.1000960
Richard,D.M.,Dawes,M.A.,Mathias,C.W.,Acheson,A.,Hill-Kapturczak, N., and Dougherty,D.M.(2009).L-Tryptophan:basic metabolic functions, behavioral research and therapeutic indications. Int. J. Tryptophan Res. 2, 45–60.
Richter,E.A.,and Ruderman,N.B.(2009).AMPK and the biochemistry of exercise:implications for human health and disease. Biochem.J. 418,261–275. doi:10.1042/BJ20082055
Rivera-Brown,A.M.,and Frontera,W.R.(2012).Principles of exercise physiology:responses to acute exercise and long-term adaptations to training. PMR 4, 797–804.doi:10.1016/j.pmrj.2012.10.007
Rivera-Chávez,F.,Lopez,C.A.,and Bäumler,A.J.(2017).Oxygen as a driver of gut dysbiosis. Free Radic.Biol.Med. 105,93–101. doi:10.1016/j.freeradbiomed.2016.09.022
Roediger,W.E.W.,Duncan,A.,Kapaniris,O.,and Millard,S.(1993).Sulphide impairment of substrate oxidation in rat colonocytes:abiochemical basis for Ulcerative colitis? Clin.Sci. 85,623–627.
Rogatzki,M.J.,Ferguson,B.S.,Goodwin,M.L.,and Gladden, L.B.(2015). Lactate is always theend produc t ofglycolysis. Front.Neurosci. 9:22. doi:10.3389/fnins.2015.00022
Rosignoli,P.,Fabiani,R.,DeBartolomeo,A.,Spinozzi,F.,Agea,E.,Pelli, M.A.,et al.(2001).Protective activity of butyrate on hydrogen peroxide-Induced DNA damage in isolated human colonocytes and HT29 tumour cells.
Carcinogenesis 22,1675–1680.doi:10.1093/carcin/22.10.1675
Ruderman,N.B.,Keller,C.,Richard,A.-M.,Saha,A.K.,Luo,Z.,Xiang,X.,et al. (2006).Interleukin-6 regulation of AMP-activated protein kinase.Potential role in the systemic response to exercise and prevention of the metabolic syndrome. Diabetes 55(Suppl.2),S48–S54.doi:10.2337/diabetes
Safdar,A.,Bourgeois,J.M.,Ogborn,D.I.,Little,J.P.,Hettinga,B.P.,Akhtar,M., et al.(2011).Endurance exercise rescues progeroid aging and induces systemic mitocondrial rejuvenation in mtDNA mutator mice. Proc.Natl.Acad.Sci. U.S.A. 108,4135–4140.doi:10.1073/pnas.1019581108
Sagar,N.M.,Cree,I.A.,Covington,J.A.,andArasaradnam,R.P. (2015).The inter play of the gut microbiome,bile acids,and volatile organic compounds. Gastroenterol.Res.Pract. 2015:e398585.doi:10.1155/2015/
398585
Sahin,E.,Colla,S.,Liesa,M.,Moslehi,J.,Müller,F.L.,Guo,M.,etal.(2011). Telomere dysfunction induces metabolic and mitocondrial compromise. Nature 470,359–365.doi:10.1038/nature09787
Sahin,E.,and DePinho,R.A.(2012).Axis of ageing:telomeres, p53 and mitochondria. Nat.Rev.Mol.Cell Biol. 13,397–404.doi:10.1038/nrm3352
Saint-Georges-Chaumet,Y.,Attaf,D.,Pelletier,E.,and Edeas,M.(2015).Targeting microbiota-mitochondriainter-talk:microbiota control mitocondria metabolism. Cell.Mol.Biol.(Noisy-Le-Grand) 61,121–124.
Saint-Georges-Chaumet,Y.,and Edeas,M.(2016). Microbiota–mitocondria inter-talk:consequence for microbiota–host interaction. Pathogens Dis. 74:ftv096.doi:10.1093/femspd/ftv096
Santhanam,S.,Venkatraman,A.,and Ramakrishna,B.S.(2007).
Impairment of mitocondrial acetoacetyl CoA thiolase activity in the Colonic mucosa of patients with ulcerative colitis. Gut 56,1543–1549. doi:10.1136/gut.2006.108449
Scott,K.P.,Gratz,S.W.,Sheridan,P.O.,Flint,H.J.,and Duncan,S.H. (2013).The influence of diet on the gut microbiota. Pharmacol.Res. 69,52–60. doi:10.1016/j.phrs.2012.10.020
Shamseer,L.,Moher,D.,Clarke,M.,Ghersi,D.,Liberati,A.,Petticrew,M.,et al. (2015).Preferred reporting ítems for systematic review and meta-analysis Protocols (PRISMA-P)2015:elaboration and explanation. BMJ 349:g7647. doi:10.1136/bmj.g7647
Shao,B.-Z.,Xu,Z.-Q.,Han,B.-Z.,Su,D.-F.,and Liu,C.(2015).NLRP3 inflammasome and its inhibitors:areview. Front.Pharmacol. 6:262. doi:10.3389/fphar.2015.00262
Shen,Z.,Ajmo,J.M.,Rogers,C.Q.,Liang,X.,Le,L.,Murr,M.M.,et al.(2009). Role of SIRT1 in regulation of LPS-ortwoethanol metabolites-inducedTNF-α Production in cultured macrophage cell lines. Am.J. Physiol.Gastrointest.Liver Physiol. 296,G1047–G1053.doi:10.1152/ajpgi.00016.2009
Shimada,K.,Crother,T.R.,Karlin,J.,Dagvadorj,J.,Chiba,N.,Chen,S.,et al. (2012).Oxidized mitocondrial DNA activates the NLRP3 inflammasome During apoptosis. Immunity 36,401–414.doi:10.1016/j.immuni.2012.01.009
Shock,L.S.,Thakkar,P.V.,Peterson,E.J.,Moran,R.G.,and Taylor,S.
M.(2011). DNA methyltransferase1,cytosine methylation,and cytosine hydroxymethylation in mammalian mitochondria. Proc.Natl.Acad.Sci.U.S.A. 108,3630–3635.doi:10.1073/pnas.1012311108
Spriet,L.L.,and Watt,M.J.(2003).Regulatory mechanisms in the interaction between carbohydrate and lipid oxidation during exercise. ActaPhysiol.Scand. 178,443–452.doi:10.1046/j.1365-201X.2003.01152.x
Steinberg,G.R.,Michell,B.J.,Denderen,B.J.W.,vanWatt,M. J.,Carey,A.
L.,Fam,B.C.,et al.(2006).Tumor necrosis factor α-induced skeletal muscle insulin resistance involves suppression of AMP-kinase signaling. Cell Metab. 4, 465–474.doi:10.1016/j.cmet.2006.
Stewart,J.B.,andChinnery,P.F.(2015).The dynamics of mitocondrial DNA heteroplasmy: implications for human health and disease. Nat.Rev.Genet. 16, 530–542.doi:10.1038/nrg3966
Storka,A.,Führlinger,G.,Seper,M.,Wang,L.,Jew,M.,Leisser,A.,etal.(2013). E. coli Endotoxin Modulates the Expression of Sirtuin Proteins in PBMCin Humans. Media.Inflamm. 2013:e876943.doi:10.1155/2013/876943
St-Pierre,J.,Drori,S.,Uldry,M.,Silvaggi,J.M.,Rhee,J.,Jäger, S.,et al.(2006). Suppression of reactive oxygen species and neuro degeneration by the PGC-1 transcriptional coactivators. Cell 127,397–408.doi:10.1016/j.cell.2006.09.024
Sugahara,H.,Odamaki,T.,Hashikura,N.,Abe,F.,andXiao,J.-Z. (2015). Differences in folate production by bifidobacteria of different origins. Biosci. Microb.FoodHealth 34,87–93.doi:10.12938/bmfh.2015-003
Taylor,R.W.,and Turnbull,D.M.(2005).Mitochondrial DNA mutations in human disease. Nat.Rev.Genet. 6,389–402.doi:10.1038/nrg1606
Trinchese,G.,Cavaliere,G.,Canani,R.B.,Matamoros,S.,Bergamo, P.,De Filippo,C.,et al.(2015).Human,donkey and cowmilk differently affect senergy efficiency and inflammatory state by modulating mitocondrial function and gut microbiota. J.Nutr.Biochem. 26,1136–1146. doi:10.1016/j.jnutbio.2015.05.003
Vermeiren,J.,VandeWiele,T.,VanNieuwenhuyse,G.,Boeckx,P.,Verstraete, W.,and Boon,N.(2012).Sulfide-andnitrite-dependent nitric oxide production in the intestinal tract. Microbial.Biotechnol. 5,379–387. doi:10.1111/j.1751-7915.2011.00320.x
Vieira,A.T.,Macia,L.,Galvão,I.,Martins,F.S.,Canesso,M. C.C.,Amaral, F.A.,et al.(2015). A role for gut microbiota and the metabolite-sensing receptor GPR43 in a murine model of gout. Arthritis Rheum. 67,1646–1656. doi:10.1002/art.39107
Vighi,G., Marcucci,F., Sensi,L., Di Cara,G., and Frati,F.(2008). Allergy and the gastrointestinal system. Clin.Exp.Immunol. 153,3–6. doi:10.1111/j.1365-2249.2008.03713.x
Viollet,B., Horman,S., Leclerc,J., Lantier,L., Foretz,M., Billaud,M., et al. (2010). AMPK inhibition in health and disease. Crit. Rev. Biochem. Mol. Biol. 45, 276–295.doi:10.3109/10409238.2010.488215
Wai,T., and Langer,T.(2016). Mitochondrial dynamics and metabolic regulation. Trends Endocrinol. Metab. 27,105–117.doi:10.1016/j.tem.2015.12.001
Wallace,D.C., Fan,W., and Procaccio,V.(2010). Mitochondrial energetics and therapeutics. Annu. Rev.Pathol. 5,297–348.doi:10.1146/annurev.pathol.4. 110807.092314
Wang,G., Yao,J., Li,Z., Zu,G., Feng,D., Shan,W.,et al.(2016). miR-34a-5p inhibition alleviates intestinal ischemia/reperfusion-induced reactive oxygen species accumulation and apoptosis via activation of SIRT1 signaling. Antioxid. Redox Signal. 24,961–973.doi:10.1089/ars.2015.6492
Wang,Y., Shi,X., Qi,J.,Li,X., Uray,K.,and Guan,X. (2012).SIRT1 inhibits the mouse intestinal motility and epitelial proliferation. Am. J. Physiol. Gastrointest. Liver Physiol. 302,G207–G217.doi:10.1152/ajpgi.00302.2011
Wei,W.,Wang,L., Zhou,K., Xie,H., Zhang,M., and Zhang,C.(2017). Rhapontin ameliorates colonic epitelial dysfunction in experimental colitis through SIRT1 signaling. Int. Immunopharmacol. 42,185–194. doi:10.1016/j.intimp.2016.11.024
Windey, K., DePreter,V., and Verbeke,K.(2012). Relevance of protein fermentation to gut health. Mol.Nutr. Food Res. 56,184–196. doi:10.1002/mnfr.201100542
Wright,D.C., Han,D.-H., Garcia-Roves, P.M., Geiger,P.C., Jones,T.E., and Holloszy,J.O.(2007).Exercise-induced mitocondrial biogénesis begins before the increase in muscle PGC-1α expression. J. Biol.Chem. 282,194–199. doi:10.1074/jbc.M606116200
Wu,Z., Puigserver,P., Andersson,U., Zhang,C., Adelmant,G., Mootha,V., et al.(1999). Mechanisms controlling mitocondrial biogénesis and respiration through the thermogenic coactivator PGC-1. Cell 98,115–124.
Xu,J., Xu,C., Chen,X., Cai,X., Yang,S., Sheng,Y., et al.(2014). Regulation of an antioxidant blend on intestinal redox status and major microbiota in early weaned piglets. Nutrition 30,584–589.doi:10.1016/j.nut.2013. 10.018
Youm,Y.-H., Nguyen,K.Y., Grant,R.W., Goldberg,E.L., Bodogai,M., Kim,D.,et al.(2015).The ketone metabolite β-hydroxybutyrate blocks NLRP3 in flammasome mediated inflammatory disease. Nat.Med. 21,263–269. doi:10.1038/nm.3804
Zaki,M.H., Boyd,K.L., Vogel,P., Kastan,M.B., Lamkanfi,M., and Kanneganti, T.-D.(2010). The NLRP3 inflammasome protects against loss of epitelial integrity and mortality during experimental colitis. Immunity 32,379–391. doi:10.1016/j.immuni.2010.03.003
Zeng,M.Y., Inohara,N., and Nuñez,G. (2017). Mechanisms of inflammation driven bacterial dysbiosis in the gut. Mucosal Immunol. 10,18–26. doi:10.1038/mi.2016.75
Zhao,L. (2010). Genomics: the tale of our other genome. Nature 465,879–880. doi:10.1038/465879a










